¶ 期刊:DEVELOPMENT
《Development》是发育生物学领域的一本老牌且具有高度影响力的国际期刊。它由 The Company of Biologists Ltd 这家非营利性组织出版,创刊于1987年(其前身可追溯至1953年的《胚胎学与实验形态学杂志》)。
¶ 技术和资源文章
¶ 跨发育过程对大脑类器官三维结构进行从介观到纳米级的成像
¶ 作者:
Juan Eduardo Rodriguez-Gatica1,‡, Vira Iefremova2,*,‡, Liubov Sokhranyaeva3, Si Wah Christina Au Yeung2,Yannik Breitkreuz4, Oliver Brüstle2,4,§, Martin Karl Schwarz3,4,§and Ulrich Kubitscheck1,§
1波恩大学物理与理论化学研究所,德国波恩市Wegeler街12号,邮编53115。
2波恩大学医学院暨波恩大学医院神经重建生物学研究所,德国波恩市Venusberg校区1号,邮编53127。
3波恩大学医学院实验癫痫与认知研究所,德国波恩市Sigmund-Freud街25号,邮编53127。
4LIFE & BRAIN股份有限公司细胞组学单元,德国波恩市Venusberg校区1号,邮编D-53127。
*现地址:美国加利福尼亚大学分子与细胞生物学系,伯克利市Weill大厅291号,邮编CA 94720
‡这些作者对本研究贡献相同
§通讯作者:brustle@uni-bonn.de;Martin.Schwarz@ukbonn.de;u.kubitscheck@uni-bonn.de
O.B. ORCID: 0000-0002-1277-0980;M.K.S. ORCID: 0000-0002-5898-751X;U.K. ORCID: 0000-0003-3750-5355
¶ 摘要
类器官是由干细胞衍生的三维培养体系,为模拟人类发育与疾病提供了新途径。脑类器官可在类组织环境中精细呈现人脑发育的多个方面,实现体外高精度研究。然而,传统光学显微镜难以观测亚细胞结构的空间关系(如远距离神经元间的突触连接)。通过快速三维超分辨率成像系统对完整类器官进行扫描可突破此局限。为此,我们开发了整合组织膨胀技术与光片荧光显微镜的成像系统,用于类器官发育过程中多种空间参数的成像与量化。该技术可在单次成像中实现从介观尺度到超分辨率的缩放,从而三维呈现细胞与亚细胞结构细节,包括清晰的有丝分裂分裂面界定以及突触前后蛋白的空间排布。我们预期光片荧光膨胀显微镜技术将推动发育与疾病相关研究中类器官的定性与定量评估。
关键词:脑类器官,膨胀显微镜,光片荧光显微镜,超分辨率,突触发生
¶ 引言
近年来,干细胞技术的进步推动了多能干细胞三维培养(如脑类器官)领域的快速发展。这类培养体系可形成自组织结构,重现体内大脑发育的某些特征。它们展现出包括顶基底面形成、极性神经上皮、神经发生性脑室区与外层放射状胶质细胞(oRG)、分层皮质样结构构建以及突触形成级成熟过程在内的早期神经发生多维度特征(Pasca等,2015;Lancaster等,2017)。然而,对这些多样化过程进行三维精准成像仍存在挑战,目前对其细胞与组织结构的分析仍主要基于类器官冷冻切片的传统二维组织学方法。值得关注的是,近期涌现的全器官透明化与成像技术通过避免冷冻切片,实现了三维结构特征的完整保留与解析,并取得显著进展(Adhya等,2021;Albanese等,2020;Edwards等,2020)。尽管已有若干研究采用全器官透明化技术,但多局限于小型类器官(如用于评估早期神经分化阶段)(Benito-Kwiecinski等,2021)。对于大型类器官的整体分析,多项参数仍具挑战性——这在类器官分化晚期出现的结构(如树突棘与突触成像)中尤为显著(Masselink等,2019;Dekkers等,2019;Albanese等,2020)。
在类器官技术发展的同时,能够三维呈现几何尺寸扩展的组织样本中精细细胞与亚细胞结构特征的新型快速大体积成像方法也应运而生。随着光片荧光显微镜(LSFM)的出现,对大范围神经元组织片段进行成像成为可能。该技术通过一或两个物镜观察荧光标记样本,其焦平面通过垂直于检测轴的薄片光进行照明(Huisken等,2004;Dodt等,2007)。因此,LSFM具备内在的光学切片能力,并可结合共聚焦线检测技术(Silvestri等,2012;Baumgart与Kubitscheck,2012)进一步提升散射样本的成像对比度。过去几年中,通过照明光束整形实现极薄光片的技术取得重大进展,从而在大视场内增强光学分辨率。晶格光片(Chen等,2014;Ellefsen与Parker,2018;Stockhausen等,2020)、艾里光束(Vettenburg等,2014)以及高斯光片腰斑视场优化技术(Dean等,2015;Fu等,2016;Neyra等,2020)相继催生了具有大扩展范围的超薄光片。通过使样本在成像物镜检测平面内沿照明光片移动,即可获取三维图像栈。若采用灵敏的高速CMOS相机,可实现每秒数百帧的成像速率,较共聚焦激光扫描显微镜显著缩短大样本的成像时间。
要实现光片显微镜的最佳成像效果,需通过使用与样本细胞组分折射率匹配的浸没介质来消除样本中的折射率不均匀性。这一目标可通过组织透明化处理实现(有关不同透明化方法的综述参见Ueda等,2020)。当采用数值孔径(NA)≥1.0的长工作距离物镜时,结合组织透明化与光片显微镜技术可实现横向约0.3μm、轴向约1.0μm的有效光学分辨率。因此,光片显微镜特别适用于快速分析大型透明化细胞簇和组织片段的复杂三维结构——例如能够对类器官的复杂三维架构进行快速光学显微镜评估(Albanese等,2020;Benito-Kwiecinski等,2021)。
然而,传统光片显微镜无法揭示神经网络的精细结构,因为这些结构远低于光学衍射极限。要在空间信息完整保留的前提下三维可视化人脑类器官中的突触,必须采用超分辨率成像技术。经典的点扫描超分辨率光学显微技术存在局限性,仅能应用于小范围区域(如单个突触),几乎无法对完整类器官乃至其内部特定结构进行三维扫描。
随着光片荧光膨胀显微镜(LSFEM)技术的发展,这一难题得以解决。该技术实现了扩展神经回路超分辨率分析(Bürgers等,2019;Gao等,2019;Schwarz和Kubitscheck,2021)。与标准膨胀显微镜技术(ExM;Chen等,2015;Ku等,2016;Tillberg等,2016;Chozinski等,2016)相同,LSFEM利用吸水性聚合物对经酶处理的组织样本进行物理膨胀。在固定组织样本内合成可膨胀聚合物前,需先用荧光抗体标记目标蛋白质抗原。随后对组织进行部分消化,使含有荧光标记的聚合物基质得以膨胀。通过膨胀处理,间距小于光学衍射极限(约250纳米)的荧光分子团得以光学分辨,从而获得类器官的有效超分辨率图像。采用LSFEM技术,我们成功实现了扩展神经回路的快速有效超分辨率成像(Bürgers等,2019)。
本研究建立了一种新型脑类器官分析流程,通过整合LSFM与LSFEM技术,实现对不同发育阶段全脑类器官的三维成像。该方法可在单个类器官样本中实现从介观到纳米级光学分辨率的无缝缩放,从而同步获取脑类器官架构与亚细胞结构的综合信息。通过精细样本制备,成功保留了常用于标记神经元亚群等结构的荧光蛋白活性。借助有效超分辨率成像技术,首次在完整透明化成熟脑类器官中实现了较大范围内神经网络参数的精细描绘,并成功识别出突触连接神经元簇。
¶ 结果
¶ 透明化处理、物理膨胀与光片显微镜技术相结合,实现了对成熟脑类器官的多维度解析
经固定的脑类器官呈现不透明结构。我们开发了特定的样本制备技术与成像方法,以最优方式提取其内在信息。为评估该方法对不同类器官制备方案的适用性,研究中采用两种方案:改编自Lancaster等(2013)的经典"类器官"方案,以及借鉴Pasça等(2015)命名的"球状体"方案。但"类器官"与"球状体"的区分具有一定主观性,因二者均基于自组织架构形成原理,故本文统称为"类器官"。我们将LSFEM技术应用于这两种方案(分别称为方案I和II,详见材料与方法),以确保广泛适用性。
固定处理后,参照Zhao等(2020)的方法,采用CHAPS替代Triton X对类器官组织进行透化处理。此举特别有利于保持自发荧光蛋白活性,并实现商业抗体对大型类器官的高效全样本染色(参见图S1)。我们常规采用DNA染色标记所有细胞核,同时使用特定细胞类型或细胞结构标志物突显特定神经元类型或亚细胞神经元结构。为实现这些样本的光学显微镜分析,透明化处理必不可少(图1A)。因此,ExM首要步骤是添加双功能连接子耦合蛋白质残基,从而在类器官内形成聚丙烯酰胺凝胶以固定荧光标记结构。蛋白酶K消化使样本透明化。将样本置于PBS等缓冲水浸介质中可引发1.5倍膨胀(图1B),而换用双蒸水介质可实现约4倍物理膨胀(图1C)。两种状态下均可使用LSFM进行分析(图1D)。通过大量对照实验证实,透明化过程未造成类器官整体结构畸变(图S2)。使用相应抗体标记紧密连接蛋白(ZO1)与祖细胞(SOX2)呈现一致结构。对比显示,透明化处理显著提升了对比度与整体图像质量。未透明类器官的不透明特性导致其内部结构无法成像。
¶ 光片荧光显微镜技术可实现对脑类器官发育过程与细胞分化进程的结构解析
¶ 荧光标记细胞亚群的追踪研究
通过透明化脑类器官的介观尺度成像,我们实现了从类器官生成到成熟的全发育过程追踪。图1E展示了类器官样本制备与成像流程的关键步骤,表1则汇总了多种成像方法。大脑类器官直径可达数毫米,为覆盖此尺度,我们采用低倍物镜(10×)与相对较低数值孔径(NA=0.3)进行光片成像,以通过有限数量的拼接图覆盖完整类器官(图1F、G)。
我们使用含10% EGFP表达细胞的混合类器官【接种前将90%诱导多能干细胞(iPSCs)与10%来自相同遗传背景的多西环素诱导EGFP标记iPSCs混合】,并通过Hoechst染色标记所有细胞核。采用优化后的样本制备方案(Bürgers等,2019;Stockhausen等,2020),通过先优化样本透化条件,再调整消化条件与膨胀缓冲液,成功保持了自发荧光蛋白(如EGFP)的荧光活性,从而无需进行抗体染色。类器官生成一个月后,从二维图像可推断EGFP细胞在整体体积中呈非均匀分布(图2A-D),这些标记细胞倾向于形成大型簇群。值得注意的是,此类EGFP标记细胞簇在培养14个月后依然存在。我们推测EGFP阳性细胞的非均匀分布源于其细胞亚群的局部增殖(图2E-G)。
¶ 光片荧光显微镜(LSFM)与光片荧光膨胀显微镜(LSFEM)可实现单一样本从介观到纳米尺度的跨维度解析
脑类器官的功能架构跨越从厘米级到纳米级的空间尺度,我们由此致力于开发一种能够覆盖此全尺度范围的记录方法。通过对透明化并经1.5倍膨胀处理的完整脑类器官使用10倍物镜进行成像(图2与图3A,B),结合4倍物理膨胀处理、1.1数值孔径物镜成像、0.3μm轴向步进扫描及后续反卷积处理,成功实现了选定样本区域100纳米级分辨率的可视化(图3C)。该方法可清晰呈现大量细胞体与延伸数百微米的神经突触结构,并支持其三维追踪。通过精细观察放大样本区域,发现众多树突棘样结构,表明神经元连接已实现高级分化与形成(图3D-F)。
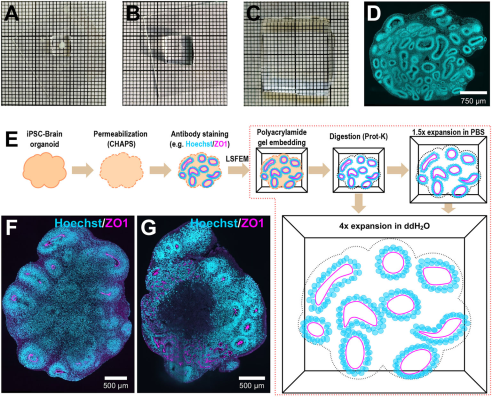 |
| 图1. 类器官样本的光片荧光膨胀显微镜制备流程。(A)包埋于聚丙烯酰胺凝胶中的两月龄脑类器官。(B)经蛋白酶K消化后的两月龄类器官,实现透明化并产生约1.5倍膨胀。(C)同一类器官在双蒸水中膨胀后实现约4倍体积扩张。(D)透明化且1.5倍膨胀类器官的光学切片,显示1.2毫米深度处的细胞核分布。(E)类器官样本制备流程示意图。三维样本固定后,使用去垢剂CHAPS进行透化处理——此为实现类器官全身免疫染色的关键步骤。随后进行特定细胞类型/结构的免疫染色及核染色。图示以紧密连接标记物ZO1(洋红色)和核染色Hoechst(青色)为例。免疫染色后将样本包埋并共价交联至聚丙烯酰胺凝胶,经含蛋白酶K的缓冲液消化使样本透明化,为光片成像创造最佳条件。消化后样本置于PBS中可产生1.5倍各向同性膨胀(对应B),而置于双蒸水中可实现4倍膨胀(对应C),从而支持全类器官的超分辨率分析。(F,G)分别展示按方案II制备的3月龄脑类器官(F)和按方案I制备的2月龄脑类器官(G)的光学切片,二者均进行了Hoechst与ZO1染色。 |
表1. 通过物理样本膨胀与特定物镜的不同组合实现多尺度脑类器官成像
| 脑类器官的光片荧光膨胀显微镜成像 | ||
| 介观尺度 (1.5倍膨胀+低倍/低数值孔径物镜配置) 感兴趣区域——完整类器官: ・类器官体积量化 ・表面积测量 ・神经管节分布模式分析 |
微观尺度 (1.5倍膨胀+高倍/高数值孔径物镜配置) 感兴趣区域——神经管节结构: ・分裂平面定位分析 ・TBR2阳性细胞/外层放射状胶质细胞量化 |
纳米尺度 (4倍膨胀+高倍/高数值孔径物镜配置) 感兴趣区域——神经突触结构: 神经突与树突棘精细结构; 突触超微架构解析 |
每个尺度均提供了感兴趣区域(ROI)的示例,并列出可能对应的图像分析参数。由于可能产生海量数据,针对特定ROI的成像在微米和纳米尺度研究中尤为重要。exp:膨胀倍数;mag:放大倍率
¶ 神经上皮结构的定性与定量评估
本研究方法能够深入解析脑类器官的细胞结构特征,因为可对众多结构参数进行系统性评估。正如我们以神经上皮玫瑰花结为例所示,该方法同样支持定量分析。当利用抗体在膨胀组织中具有优异渗透性这一特性时(Edwards等,2020),结构分析效能可得到进一步提升。
这些结构典型存在于大脑类器官中,形成类脑室区(VZ)结构域,其顶面可通过紧密连接蛋白ZO1(亦称TJP1)抗体进行标记。ZO1免疫荧光从而能够评估神经上皮玫瑰花结的拓扑结构及其可能包绕的类脑室腔隙(图4A,B)。图4C展示了整个类器官中裁剪出的顶面结构,显示这些结构在尺寸和形态上存在显著变异:部分为封闭结构(即包绕类脑室腔隙),另一些则呈现相对扁平形态,具有复杂几何特征和片层状拓扑结构。因此,测量神经上皮顶面面积成为其表征的理想参数。图4A所示类器官分割结构的表面尺寸分布展示于图4C。通过图4C所示数据可实现多参数量化分析(见表1),例如顶面拓扑结构特征化(图4D)。通过对三个独立2月龄类器官进行成像,成功量化了介观尺度参数。
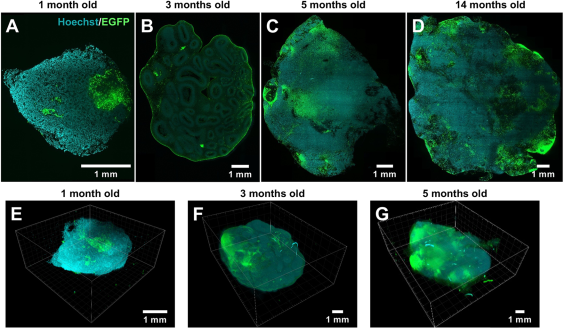 |
| 图2. 含10% EGFP表达细胞的嵌合脑类器官在14个月时间跨度内的发育过程。(A-D)经透明化与1.5倍膨胀处理的脑类器官光片荧光显微镜图像,显示培养1个月(A)、3个月(B)、5个月(C)及14个月(D)后EGFP表达细胞(绿色)与Hoechst核染色(青色)的分布。所有类器官均按方案I制备。所示光学切片深度分别为646µm、1140µm、1830µm和1119µm,图像尺寸依次为3.6×3.6 mm²、8.6×11.7 mm²、13.1×14.9 mm²和16.6×17.3 mm²。(E-G)分别为1月龄(E)、3月龄(F)和5月龄(G)脑类器官的三维重构图像,成像体积对应为3.6×3.6×1.6 mm³、8.6×11.7×4.4 mm³和13.1×14.9×5.2 mm³。图2B的Hoechst通道展示于图1D用以说明膨胀过程。 |

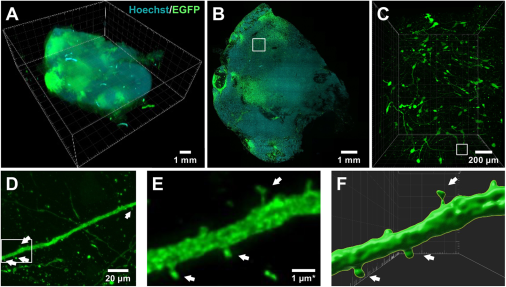 |
| 图3. 按方案I制备的5月龄含GFP阳性细胞脑类器官,呈现从厘米级到纳米级的多尺度成像。(A)三维重构图像,体积为13.1×14.9×5.2 mm³。(B)深度1.8 mm处的光学切片,该图像使用10×/0.3 NA物镜获取,并作为类器官时序发育系列图的一部分展示于图2C,尺寸为13.1×14.9 mm²。(C)对B图中白色框标区域(体积1248×1548×1275 µm³)进行的三维图像栈渲染,该图像在4倍膨胀后使用25×/1.1 NA物镜对同一样本采集。(D)C图中框选区域放大(185×132 µm²),显示树突棘样结构(箭头指示)。(E)D图中框选区域进一步放大,经校正的标尺1 µm*已考虑4倍膨胀效应,实际物理尺寸对应4 µm。(F)神经突触结构的表面渲染图清晰呈现树突棘形态。C-E所示图像数据均经过反卷积处理。本次实验共采集35个图像栈,覆盖样本区域1248×1548 µm²,总深度1275 µm³,轴向步进精度为0.3 µm。 |

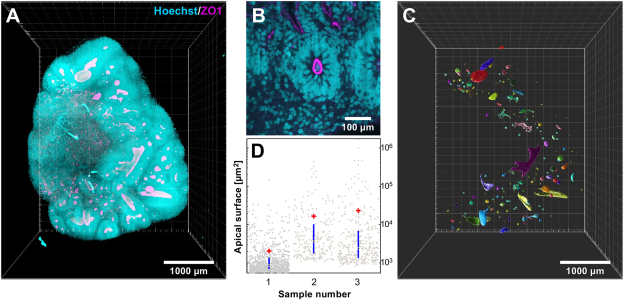 |
| 图4. 使用ZO1抗体对2月龄脑类器官神经上皮结构顶面进行标记(参见视频1)。通过对三个独立2月龄类器官进行成像与评估,获得如下定量参数。所有类器官均按方案I制备并在PBS溶液中测量。(A)细胞核(Hoechst染色,青色)与ZO1(洋红色)分布。青色荧光细胞核标示类器官大致形态,由此获得总体积(1.36±0.55)×10¹⁰ µm³ 和总表面积(1.2±0.6)×10⁸ µm²。(B)深度1 mm处光学切片放大显示封闭式顶面结构,可见玫瑰花结内部类脑室区腔隙。鉴于每个玫瑰花结包含一个顶面,统计三个完整类器官平均包含485±270个玫瑰花结。(C)图A类器官内部神经上皮顶面结构的裁剪展示,颜色根据表面积大小随机生成。(D)三个类器官顶面面积分布情况(因非所有结构均形成封闭容积),该参数可作为特征化表征指标。红色十字标示平均值,蓝色线条表示四分位距,黑点显示中位数。整体平均值为(1.66±4.79)×10⁴ µm²,中位数为3710 µm²。 |
¶ 神经亚群的界定
抗体染色与光片荧光膨胀显微镜(LSFEM)技术的结合,为神经亚群的直接检测提供了有效手段。以SOX2与TBR2(EOMES)抗体的共标记为例:该策略可特异性界定外层放射状胶质细胞(oRG)——这类位于脑室外下层(oSVZ)的特殊神经元祖细胞群体在人类大脑发育中至关重要,负责神经发生与皮质扩张(Bershteyn等,2017)。oRG细胞的典型特征是在脑室区(VZ)外层表达SOX2而不表达TBR2(详见图5A-C绿色箭头指示)。
然而,仅通过SOX2阳性细胞及其定位并不足以实现oRG细胞的明确界定与量化(Pollen等,2019)。最新研究表明,绝大多数表达HOPX的细胞同时表达放射状胶质细胞标志物SOX2,但不表达中间祖细胞标志物TBR2(Bhaduri等,2020;Pollen等,2019)。因此,HOPX被视为oRG细胞的有效标志物,我们将其应用于3月龄类器官研究。图6展示了不同样本中oRG细胞识别的多个范例。通过与N-钙黏蛋白抗体的共染色,成功标定了神经上皮顶面结构,进而揭示了HOPX阳性细胞与组织架构的空间关系(图6C,D)。
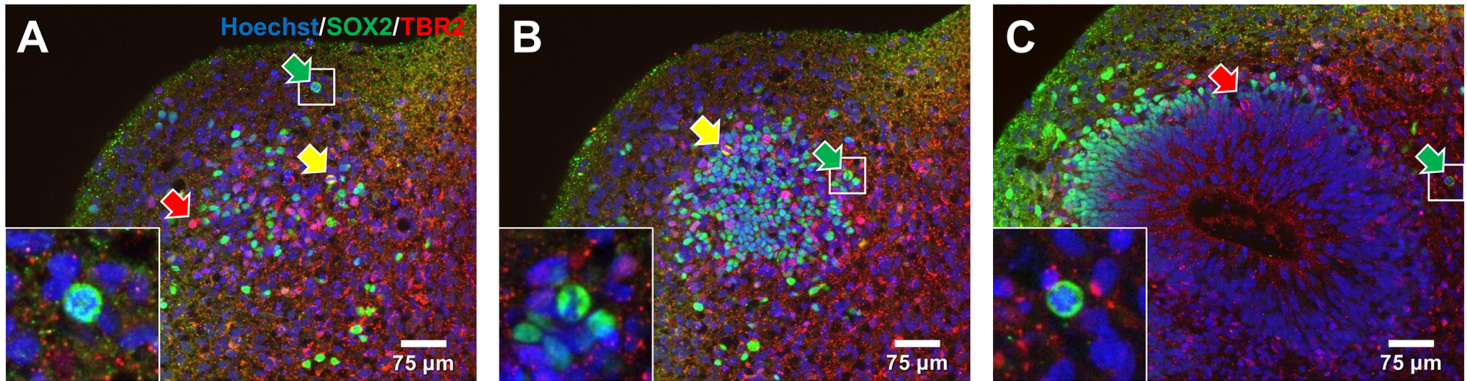 |
| 图5. 采用SOX2(绿色)与TBR2(红色)抗体双标记法在2月龄类器官(方案I,PBS溶液中Hoechst核复染蓝色)中鉴定不同皮质祖细胞类型(oRGs)。(A-C)同一样本在53µm(A)、75µm(B)和146µm(C)深度获取的单层光学切片。分别标记了TBR2阳性/SOX2阴性(红箭)、TBR2与SOX2双阳性(黄箭)及TBR2阴性/SOX2阳性(绿箭)细胞。此类基底区分布的TBR−/SOX2+细胞提示为oRG细胞(Pollen等,2019)。总观察区域:703×651µm²。插图显示框选区域的放大视图。 |
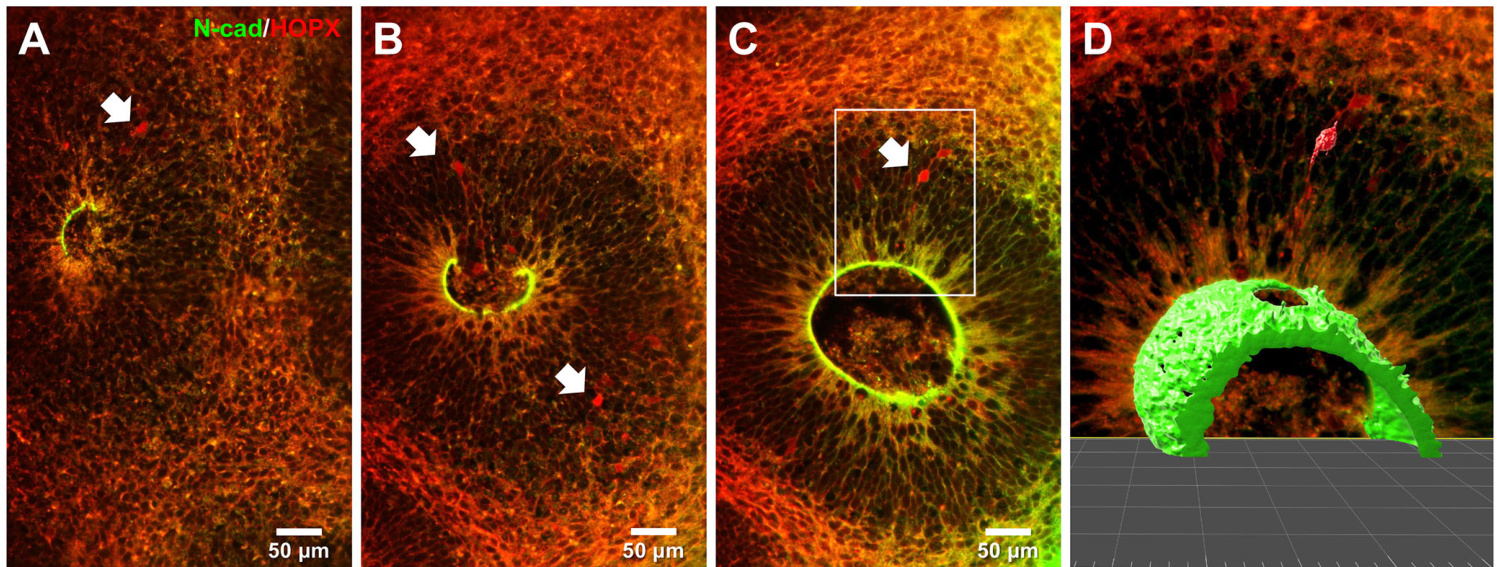 |
| 图6. oRGs细胞相对于脑室区(VZ)表面的三维空间定位界定。(A-C)经N-钙黏蛋白(N-cad,绿色)与HOPX(红色)标记的3月龄脑类器官,白色箭头指示oRGs细胞。类器官按方案I制备并在PBS溶液中测量。A-C分别显示深度200µm、452µm和595µm处3µm厚度光学切片的最大强度投影。(D)C图标注区域的oRGs与VZ结构表面三维重构。 |
¶ 亚细胞结构成像
¶ 有丝分裂分裂平面取向
人脑发育过程中神经发生的一个关键参数,是神经元祖细胞有丝分裂分裂平面相对于神经上皮顶面的取向(图7A)。有丝分裂纺锤体的取向调节分裂平面的方向,从而决定两个子细胞的位置。在人类皮质发生早期阶段,正确的纺锤体取向对于实现对称与非对称细胞分裂的适当比例至关重要。在皮质早期发育过程中,大多数活跃分裂的神经元祖细胞表现出相对于脑室表面分裂平面的水平取向(即0-30°角),通过对称性细胞分裂促使皮质祖细胞池扩增。而垂直(60-90°)和斜向(30-60°)的有丝分裂分裂平面在神经发生前夕开始占据主导(LaMonica等,2013;Yingling等,2008)。这种不对称细胞分裂模式会产生两个不同的子细胞,并促进神经元分化。多项研究表明,有丝分裂分裂平面取向的紊乱会导致皮质发生异常,并表现为多种发育表型。Lancaster团队开展的里程碑研究表明,有丝分裂分裂平面取向的转变对小头畸形的发育具有关键作用(Lancaster等,2013)。最近两项使用Miller-Dieker综合征患者来源类器官与对照组对比的研究进一步证实:在疾病条件下,放射状胶质细胞的细胞分裂平面从垂直向水平明显转变,而斜向平面未显著增加,从而导致神经发生提前且患者来源的3D皮质培养物体积缩小(Bershteyn等,2017;Iefremova等,2017)。
我们发现,通过Hoechst DNA染色结合ZO1与神经祖细胞标志物SOX2的免疫荧光染色,可有效界定分裂平面相对于神经上皮表面的取向(图7B,C)。重要的是,图像分析表明仅依靠二维投影无法准确评估分裂平面相对于顶面的取向。如图7D所示,xy平面投影提示该有丝分裂细胞的分裂平面相对于顶面呈水平取向,而同一细胞核的xz平面投影却显示垂直取向(图7E)。真实取向的评估需依赖完整的三维视图(图7F)。我们对三个独立2月龄脑类器官进行了此项分析,结果如图7G所示。分析表明,沿类脑室结构分布的分裂细胞中多数(53%)呈现垂直分裂平面取向,这与既往皮质类器官培养早期阶段的研究数据一致(Bershteyn等,2017;Iefremova等,2017)。
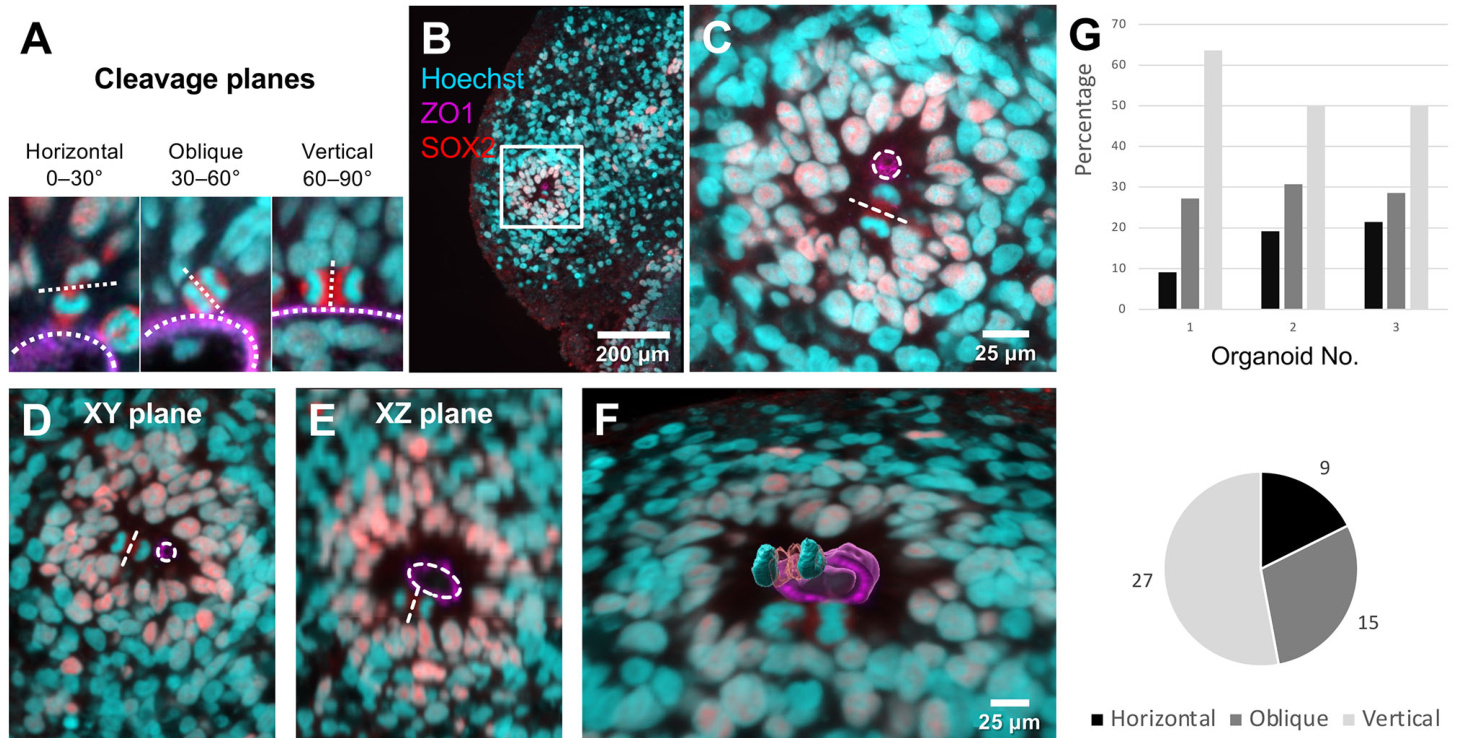 |
| 图7. 分裂平面分析。(A)分裂平面相对于脑室区顶面腔隙表面的定义。(B)采用方案I制备的2月龄脑类器官经ZO1(洋红色)、SOX2(红色)和Hoechst(青色)标记,ZO1染色显示脑室区腔隙表面。总观察区域:。(C)B图中框选区域放大,可观察有丝分裂细胞分裂平面(虚线)相对于腔隙表面的取向。A、C-E中虚线圆标示神经玫瑰花结的顶面。总观察区域:。(D,E)仅使用二维数据可能导致分裂平面取向的误判。(F)真实分裂平面取向仅能从三维数据中推导(视频2)。(G)基于三维数据对三个独立2月龄类器官分裂平面取向的量化分析。下图显示各取向占比及数量总和。 |
¶ 突触前后结构的检测与空间定位关系
此前研究已报道过大脑类器官中存在功能性神经元连接(Pasca等,2015;Quadrato等,2017;Giandomenico等,2019)。然而,目前尚未有研究利用完整透明化类器官,以空间方式展示突触中突触前与突触后蛋白的共存现象(当前类器官成像技术综述参见Brémond Martin等,2021)。我们再次使用含10% EGFP细胞的嵌合类器官研究此类结构。图8显示一个直径约1.5厘米的14月龄类器官(图8A),其近表面区域包含大量具有神经元形态和长神经突的细胞(图8B)。通过针对突触前蛋白synapsin1(SYN1)和突触后蛋白HOMER1的光片荧光膨胀显微镜(LSFEM)分析,发现沿神经投射分布的点状信号间距为150±61纳米(平均值±标准差;n=26),中位值为130纳米(图8C-F)。在该类器官中,我们测量了三段不同树突的棘密度:经MAP2抗体标记的膨胀类器官显示,长度分别为269μm、187μm和175μm的树突段上,棘密度分别为0.06个/μm²、0.036个/μm²和0.045个/μm²,相当于平均每212μm²出现一个树突棘。结合上述突触前后结构的证实,这些数值表明14月龄类器官已形成发育完善且丰富的神经元连接。因此,LSFEM技术可在无需物理切片的情况下,在完整三维类器官中检测树突棘结构(图3D-F)和突触结构(图8D,E)。
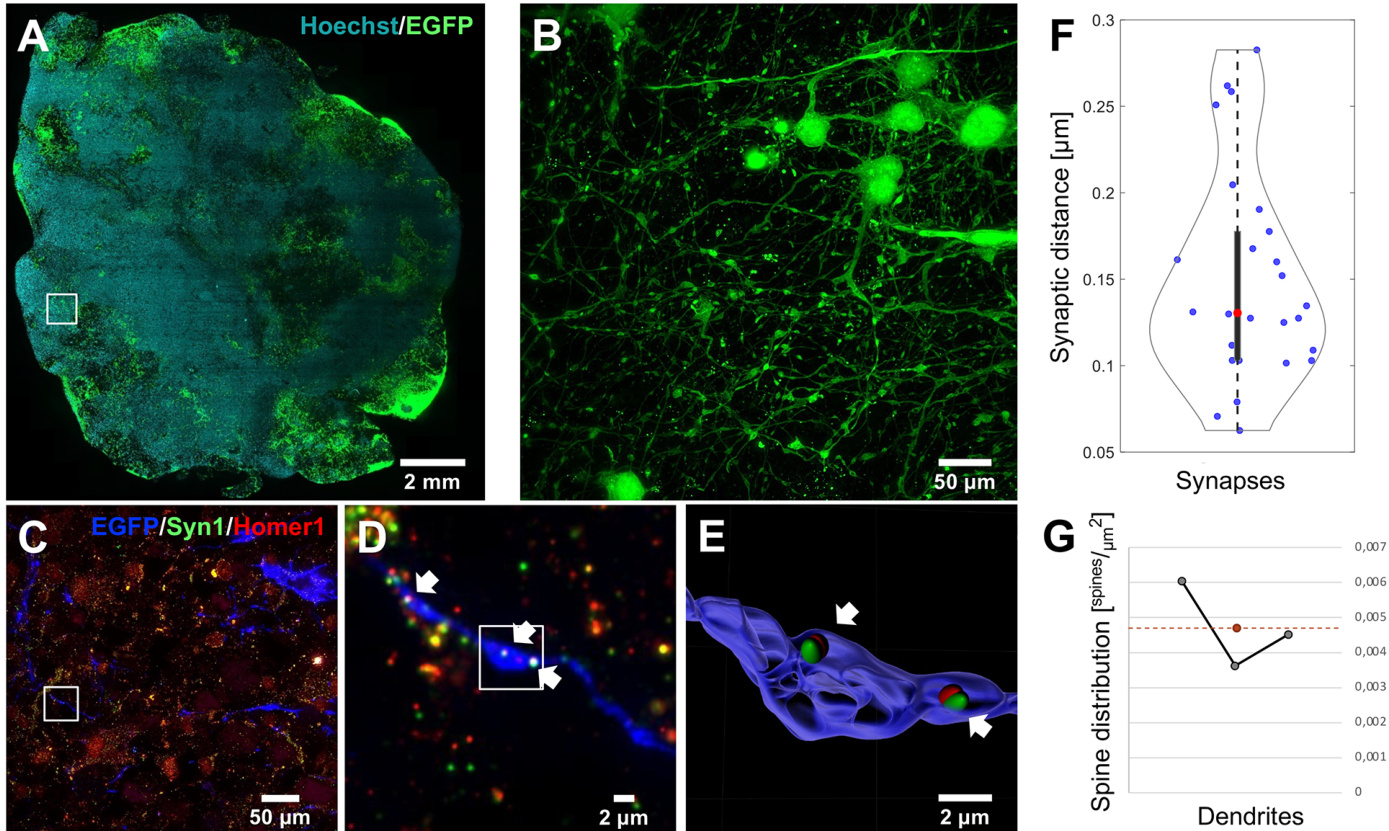 |
| 图8. 按方案I制备的14月龄脑类器官中的突触前后结构。(A-G)该类器官含10% EGFP表达细胞,经Hoechst核染色及突触前蛋白synapsin1(SYN1,绿色)与突触后蛋白HOMER1(红色)抗体标记。详见视频3。(A)深度1.5 mm处光学切片,通过285个单幅图像拼接构建,使用10×/0.3 NA水浸物镜采集,该图像亦作为类器官发育时序系列图部分内容展示于图2D,尺寸16.6×17.3 mm²。(B)经4倍膨胀及反卷积处理后,使用25×/1.1 NA物镜采集1000张图像(轴向范围约300µm)的最大强度投影,尺寸547×547 µm²。(C)20张图像(轴向约300µm)的最大强度投影,其中EGFP显示为蓝色,突触前后蛋白SYN1和HOMER1分别显示为绿色与红色。(D)C图中框选区域放大显示突触前后蛋白SYN1与HOMER1沿轴突终末的共定位,箭头指示神经突沿线的突触结构。(E)D图中框选区域多突触终末的三维重构。(F)三维分析中突触前后标记物间距的小提琴图(蓝点),红点表示中位数,黑线为四分位距,虚线为须线范围。(G)三段不同树突的棘密度分析。红色标记平均密度,对应每212µm²一个树突棘(0.0047个/µm²)的平均值。 |
¶ 讨论
脑类器官是尺寸达数毫米级的不透明三维结构。其不透明特性会阻碍传统成像获得足够对比度,而体积因素则限制了高分辨率光学显微镜的应用——因为高数值孔径(NA)物镜通常具有极短的工作距离。使用类器官切片的传统成像方法会丢失这类复杂样本固有的三维特征。
本研究证明了光片荧光显微镜(LSFM)结合1.5-4倍类器官透明化膨胀技术的潜力(该技术近期亦应用于小鼠脑切片研究,称为LSFEM;Bürgers等,2019;Gao等,2019;Stockhausen等,2020)。实验表明该方法可对直径达15毫米的类器官进行精细分析。
膨胀过程中自发荧光蛋白活性的保留,使我们能够追踪特定细胞类型在长达14个月发育周期中的定位与命运轨迹。通过在构建类器官时使用部分表达荧光蛋白的细胞并结合核染色,实现了对细胞群在三维空间内分布规律的深度解析。由此我们证实了已知现象:类器官的发育会因批次间差异性而存在显著变异(Quadrato等,2016,2017;Qian等,2019;Velasco等,2019)。
¶ 跨尺度成像
神经元连接的关键细节发生在约100纳米尺度的空间范围内。如此微小的结构只能通过超分辨率光学显微镜进行解析。我们已证实光片荧光膨胀显微镜(LSFEM)可实现横向低于100纳米、轴向达300纳米的有效超分辨率成像, 因此能够识别单个突触连接(Bürgers等,2019)。实现此目标需对样本进行透明化处理随后四倍物理膨胀及后续高分辨率成像。通过反卷积技术还可进一步提升图像分辨率。该方法使我们能在5月龄脑类器官中检测到单个树突棘样结构。通常,实现如此高分辨率的成像在技术层面可行,但由于会产生海量数据,难以应用于大体积类器官样本。一个原始体积为1立方毫米的类器官经膨胀后体积将达64立方毫米。鉴于奈奎斯特定理,以16位深、横向100纳米/轴向300纳米的分辨率对该结构成像,将产生340TB的数据集。当前数据处理工作站处理如此规模数据时已达或超出其计算极限。因此,我们采用介观尺度成像定位特定区域,进而获取该区域的超分辨率数据。值得注意的是,这种方法可在单一样本上实现从1厘米到100纳米(跨越五个数量级)的跨尺度观测。
¶ 介观尺度
发育中脑类器官的一个典型特征是生成具有顶基极性的玫瑰结状神经上皮结构。这些结构包绕的空间可形成类脑室系统,其范围可跨越类器官的大部分区域(Di Lullo和Kriegstein,2017)。由于这些区域对应于体内神经发生性脑室区的对应结构,对其进行定性与定量评估具有重要意义。使用LSFEM技术研究这些结构具有显著优势——它们在我们获取的三维数据中可直接呈现。通过对ZO1或N-钙黏蛋白的附加染色,能够清晰可视化并分析脑室区的腔隙结构。
¶ 微观尺度
脑类器官结构的另一关键决定因素是外层放射状胶质细胞(oRGs)。这类细胞可通过TBR2、SOX2和HOPX抗体进行标记(Pollen等,2019)。由于oRGs在脑类器官中丰度较低,其鉴定通常具有挑战性。若仅采用二维切片会使问题更加复杂——因为测绘体积十分有限。使用完整三维图像栈显著提高了检测这一重要细胞类型的成功率。为实现高质量对比度标记,在标记前采用新型透化策略至关重要:即使用CHAPS替代Triton X(Zhao等,2020)。该方法使抗体在成熟大型类器官内部靶点的渗透效率显著提升。分裂细胞分裂平面相对于脑室区(VZ)表面的取向对脑类器官生长特性具有重要意义。我们发现在三维数据中能准确评估分裂平面相对于腔隙表面的取向——与仅使用二维光学切片相比,三维数据可避免可能的误判。更重要的是,本方法仅需使用SOX2即可实现分裂平面取向的可视化,无需采用磷酸化波形蛋白等特殊抗体。
¶ 纳米尺度
显然,光片荧光膨胀显微镜(LSFEM)在分辨率和样本精细结构检测方面无法与电子显微镜媲美。然而,与电子显微镜相比,LSFEM具有多色荧光成像兼容性,可在单次大组织制备中同时实现不同神经元群体的分子对比度与纳米级分辨率。我们采用EGFP联合突触前后标记物对脑类器官进行三色染色,从而精确识别突触结构。通过LSFEM技术,我们首次在14月龄类器官中成功检测到沿神经投射分布的突触前synapsin1与突触后HOMER1蛋白(间距约150纳米),以光学方法明确证实了突触的存在。这种多尺度成像能力使得特定脑区突触的数量统计与空间分布分析成为可能。原则上,本技术提供的光学分辨率足以在三维空间中识别不同种类的树突棘(如蘑菇棘或钝棘)。未来此类实验可采用MAP2标记树突结构以增强解析度。
需要指出的是,LSFEM存在特定局限性:其显著特征是非各向同性分辨率——轴向分辨率通常比横向分辨率低约三倍。虽存在改善该差异的技术方案,但它们无法在极限分辨率下生效。此外,对任意尺寸样本进行超高分辨率成像并非易事,部分原因在于抗体在空间扩展样本中的渗透时间随样本厚度呈非线性增长,因此实现大样本均匀染色具有挑战性。同时,高分辨率成像物镜有限的工作距离也限制了超厚样本的观测。
总而言之,我们证实当LSFM与ExM结合形成LSFEM技术,并与保留自发荧光蛋白的精细样本制备方案及反卷积优化技术相结合时,可在单次成像中实现从完整成熟脑类器官到突触级分辨率的多尺度成像。利用抗体在膨胀组织中具有优异渗透性这一特性(Edwards等,2020),成像效果还可进一步提升。因此,LSFEM堪称脑类器官发育分析的理想技术平台。
¶ 材料与方法
¶ 多能干细胞培养
本实验所用人诱导多能干细胞(iPSCs)为细胞系iLB-C-133bm-s4(hPSCreg注册号UKBi013-A)及其衍生细胞系iLB-C-133bm-s4 AAVS1-GFP(hPSCreg注册号UKBi013-A-1)。细胞维持培养于经1% Geltrex膜基质(Thermo Fisher Scientific)包被的六孔组织培养板(Nunc)中,培养体系为StemMACS iPS-Brew(Miltenyi Biotec),并使用EDTA/PBS溶液进行常规传代。所有培养物均经过支原体检测且始终保持无支原体状态。
¶ iPSC来源三维类器官的制备
¶ 方案1(基质胶+轨道摇床)
类器官制备参照既往已建立方案并稍作修改(Iefremova等,2017)。简要流程如下:在类器官培养第0天,使用TrypLE Express(Gibco)将iPSCs消化为单细胞悬液,随后在超低吸附圆底96孔板每孔接种18,000个细胞,培养体系为含50 μM ROCK抑制剂Y-27632(10 μM,Hiss Diagnostics)的StemFlex培养基(Gibco)。为可视化EGFP阳性细胞,将携带与未携带多西环素诱导型EGFP构建体的同遗传背景iPSCs按10:90比例混合。类器官隔天更换StemFlex培养基培养至第5天,随后转移至低吸附6cm培养皿,改用含50% Neurobasal与50% DMEM/F12的神经诱导培养基(补充1× N-2、B-27补充剂(均购自Gibco)及葡萄糖(0.4 mg/ml,Carl Roth))。换液前向神经诱导培养基中添加1% MEM非必需氨基酸溶液(MEM-NEAA,Gibco)、1% GlutaMax、LDN-193189(180 nM,Axon Medchem)、A8301(500 nM,Miltenyi Biotec)和XAV939(10 μg/ml,Enzo Life Sciences)。培养5-6天后更换为神经分化培养基(含50% Neurobasal与50% DMEM/F12,补充1× N-2、B-27、葡萄糖(0.4 mg/ml)、cAMP(0.15 μg/ml,Sigma-Aldrich)、1% MEM-NEAA、1% GlutaMax)。随后5天内将类器官包埋于Matrigel(Corning Life Sciences)中,并在细胞培养摇床上继续培养,每2-4天换液直至固定处理日。从第35天起更换培养基为含50% Neurobasal与50% DMEM/F12(补充1× N-2、1× B-27、葡萄糖(0.4 mg/ml)、cAMP(0.15 μg/ml)、胰岛素(Sigma-Aldrich)、1% Matrigel、20 ng/ml脑源性神经营养因子(BDNF;CellGS)及10 ng/ml胶质细胞源性神经营养因子(GDNF))。
¶ 方案II(CERO 3D细胞培养),采用改良Pasca方案
¶ 实验前准备
细胞准备: 准备处于良好生长状态的iPSCs。
材料准备: 配制好所需的各种基础培养基和添加剂。
¶ 第1步:制备单细胞悬液与接种 (Day 0)
消化解离: 使用 StemPro Accutase 酶(一种温和的细胞解离酶)将贴壁生长的iPSCs消化下来,并吹打成单细胞悬液。
细胞计数: 对细胞进行计数。
混合细胞 (可选): 根据实验设计,将携带诱导型eGFP报告基因的iPSCs与未携带的iPSCs以 10% : 90% 的比例混合。此举通常是为了便于后续追踪特定细胞群的命运。
接种至微孔板: 取 150万 个细胞,接种到 AggreWell 800 微孔板中。该板的设计使得细胞会聚集在每个微孔底部,形成均匀的细胞团。
配制初始培养基: 所使用的培养基是为神经分化设计的,成分复杂,旨在提供最佳的营养和信号环境。培养基中还添加了:
两种 SMAD通路抑制剂 (Dorsomorphin 和 SB-431542): 启动神经分化。
ROCK抑制剂 (Y-27632): 提高刚解离的单细胞存活率。
¶ 第2步:聚集形成与早期分化 (Day 1 - Day 5)
培养: 将接种好的AggreWell板放入培养箱中静置培养。
换液: 每天更换一次新鲜的培养基。注意: 此阶段的新鲜培养基不再添加ROCK抑制剂(Y-27632),因为细胞团已初步形成,不再需要它来防止凋亡。但SMAD抑制剂仍需保留。
¶ 第3步:转移至生物反应器与扩增 (Day 5)
转移: 在第5天,将初步形成的细胞团(此时可称为“胚体”或早期类器官)从AggreWell板中取出,转移至 CERO培养管 中。
生物反应器培养: 将CERO培养管放置于 旋转式CERO台式生物反应器 上,开始动态悬浮培养。旋转系统使类器官保持悬浮,生长得更好更大。
换液频率: 从第5天到第12天,每两天(隔天) 更换一次培养基。培养基成分与此前相同(含SMAD抑制剂,不含ROCK抑制剂)。
¶ 第4步:分化诱导 (Day 12 - Day 16)
更换分化培养基: 从第12天开始,更换培养基配方。
关键改变: 新培养基去除了SMAD通路抑制剂,并添加了 bFGF(碱性成纤维细胞生长因子)。
目的: bFGF是促进神经前体细胞存活、增殖和分化的关键生长因子。此步骤旨在引导类器官进一步向特定的神经谱系分化。
¶ 第5步:成熟与维持 (Day 16 以后)
更换维持培养基: 从第16天开始,使用无任何特殊补充剂(即不含SMAD抑制剂和bFGF) 的基础培养基。
目的: 让类器官在更接近基础的环境中进行长期的成熟和自组织。
换液频率: 每两天(隔天) 更换一次维持培养基,直至实验结束。
¶ 制备含多西环素诱导型EGFP标记iPSCs的混合三维培养物
为根据方案I或II制备含EGFP细胞的混合三维皮质类器官,我们采用携带多西环素诱导型EGFP表达框的iPSCs(该构建体通过基因敲入技术整合至AAVS1基因座,具体方法见Qian等(2014)和Peitz等(2020))。使用StemPro Accutase酶(Gibco)将iPSCs消化为单细胞悬液,随后将90%未标记iPSCs与10%同遗传背景的EGFP标记iPSCs轻柔充分混合,按前述方法接种至AggreWell板中。从混合培养第0天起,每次换液时持续添加多西环素(1 μg/ml,Sigma-Aldrich)。除多西环素处理外,三维培养条件与含100%未标记细胞的iPSC衍生培养物保持完全一致。
¶ 样本制备与显微镜技术
本节概述样本制备、膨胀处理及显微镜检测的整体方法。具体短期与延长方案的详细实验步骤及所需溶液配置,请参见补充材料与方法部分。
¶ 免疫化学
免疫组化方案基于标准流程进行优化,所用全部化学试剂汇总于表S1,具体操作细节详见补充材料与方法部分。简要流程如下:固定后的三维培养物首先在透化缓冲液(1× PBS, 0.5% CHAPS)中于37°C摇床上进行透化处理,处理时间根据样本尺寸调整(例如1月龄样本需1小时)。透化后样本用1× PBS在室温(RT)下清洗三次。为阻止一抗非特异性结合,样本在封闭缓冲液(1× PBS, 5%正常山羊血清, 0.3% Triton X-100, 0.02%叠氮化钠)中于室温摇床上过夜(ON)孵育。封闭完成后,类器官在含一抗(参见表S2)的封闭缓冲液中于4°C摇床上过夜孵育。次日,切片在室温用封闭缓冲液清洗三次(每次30分钟),随后在二抗(参见表S3)中于4°C摇床上过夜孵育。所有样本均使用Hoechst 33342(H3570, Invitrogen)进行核染色。
¶ 类器官膨胀技术
本实验采用基于Chozinski等(2016)的膨胀显微镜方案,具体操作细节详见补充材料与方法部分。简要流程如下:免疫染色后的类器官与2 mM甲基丙烯酸-NHS连接剂在室温摇床上孵育24小时。PBS清洗三次后,将类器官置于单体溶液(含8.6%丙烯酸钠、2.5%丙烯酰胺、0.15% N,N'-亚甲基双丙烯酰胺、11.7% NaCl的1× PBS溶液)中,于4°C摇床上孵育16小时。
凝胶溶液通过向新鲜单体溶液中添加4-羟基-TEMPO(0.01%)、TEMED(0.2%)和过硫酸铵(0.2%)配制。凝胶过程中,类器官置于24孔板中冰浴以防止提前聚合。加入凝胶溶液后,样本在4°C摇床上震荡5分钟,随后转移至凝胶反应室,37°C孵育3小时。凝胶形成后,将样本置于消化缓冲液(50 mM Tris、1 mM EDTA、0.5% Triton X-100、0.8 M盐酸胍、16 U/ml蛋白酶K;pH 8.0)中37°C孵育,每24小时更换缓冲液。通常而言,2月龄类器官需约2个完整日方可完全消化。消化完成后移除缓冲液,用PBS清洗样本三次。
¶ 光片显微镜技术
本研究采用定制搭建的光片显微镜系统。简要而言,荧光激发采用四组光纤耦合激光器(Hübner Photonics),发射波长分别为405 nm、488 nm、561 nm和638 nm。通过镀银振镜系统生成水平扫描光片,样品室内光束腰位置的调节通过线性精密平台上的中继光学系统实现。物镜面光束腰1/e²直径调整为:405 nm激光6.5±0.02 μm、488 nm激光7.3±0.02 μm、561 nm激光7.0±0.02 μm、638 nm激光8.3±0.02 μm。照明系统采用Mitutoyo 10×/0.28 NA空气物镜。定制样品室设有由常规24×24 mm盖玻片(厚度0.17 mm)形成的照明窗,样本通过不同物镜从顶部进行观测(详见表2)。样本固定于盖玻片上,可通过电动微位移台实现三维空间移动。部分实验选用1.5倍放大模块(Nikon)。数据采集采用sCMOS相机(2048×2048像素,像素尺寸6.5 μm,Orca Flash 4.0 V2,滨松光子学株式会社),全局快门模式工作。所有电子组件均通过自主编写的LabView程序控制。
¶ 介观尺度成像
介观尺度成像与分析可获取完整类器官的拓扑结构信息,有助于分析批次间差异性等应用。
成像时,消化后的样本经多聚赖氨酸固定于盖玻片上以防止测量过程中位移。随后将盖玻片插入样品架并置于充满PBS溶液的样品室中,实现1.5倍膨胀。图像采集前进行视觉检查以确认样本制备成功,继而使用10×水浸物镜(NA 0.3,有效视场998μm²)通过光片荧光显微镜(LSFM)进行分析。实际达到的有效光学分辨率参数见表2。由于样本尺寸较大,需采用镶嵌式成像方式以覆盖整个类器官。
¶ 微观尺度成像
对于微观尺度成像,类器官按上述方法制备成透明化的1.5倍膨胀样本,此时采用数值孔径(NA)1.1的高分辨率长工作距离物镜进行观测,可获得约0.3μm横向和1.1μm轴向的光学分辨率。考虑样本膨胀效应后,实际有效分辨率分别达到0.2μm和0.7μm(表2)。与介观成像相比,横向分辨率提升约4倍,轴向分辨率提升约15倍,从而实现细胞尺度的结构表征。在此分辨率下对完整类器官成像,每1mm³样本每通道将产生约500GB数据,需高端图像处理工作站进行分析。因此尽管技术可行,通常不建议在此分辨率下进行完整类器官成像,而应在介观数据中选定特定感兴趣区域(ROIs)进行后续微观尺度分析。
¶ 纳米尺度成像
对于纳米尺度成像,类器官按上述方法制备成透明化的1.5倍膨胀样本后,将样本所处缓冲溶液更换为双蒸水,从而实现相较于原始样本尺寸约4倍的膨胀。此类样本采用高分辨率长工作距离物镜(NA 1.1)进行检测,可获得约0.3μm横向和1.1μm轴向的光学分辨率。考虑样本膨胀效应后,实际分别达到0.1μm和0.3μm的有效超分辨率(表2),从而实现亚细胞尺度的结构表征。通过三维图像反卷积处理可进一步提升分辨率。成像时需调整轴向步长以确保反卷积效果最优化。
¶ 数据处理
我们采用自主编写的MATLAB脚本处理16位原始图像的三维图像栈,实现并行数据处理(Gonzalez等,2009)。首要步骤是通过调整强度直方图,使整个数据集的亮度和对比度标准化。
使用Fiji(Schindelin等,2012)及其拼接插件(Preibisch等,2009)将多个三维数据集拼接后,可获得样本的完整三维重构。为优化拼接过程(特别是在数据量超过工作站内存时),采用两步处理法:首先通过Fiji脚本创建三维数据集的子栈,每个子栈包含原完整栈中心区域约15%的信息;随后根据互相关度量最佳重叠原则,将各子栈与其相邻子栈进行拼接。基于拼接后各子栈的定位信息,最终完成全三维图像栈的拼接。
拼接后执行最终对比度优化步骤,以补偿样本在轴向可能的强度差异。对此采用直方图均衡化处理拼接数据集的每个图像平面。Z轴投影计算使用Fiji的最大强度投影算法实现。
¶ 反卷积处理
如结果部分所述,选用Huygens软件(专业版21.10,Scientific Volume Imaging)对选定图像栈进行空间反卷积处理。基于显微镜参数采用理论点扩散函数(PSF),或通过分析1%琼脂糖凝胶中荧光微珠测定实测PSF进行反卷积。使用经典最大似然估计算法,信噪比设定为12-20,最大迭代次数设为60-100次。
通过Imaris软件(版本9.7.2,Bitplane)的Surpass视图实现数据三维重构。数据处理在工作站上完成,其配置包括:两颗英特尔至强铂金8160处理器(2.1 GHz,24核心)、512 GB内存及英伟达Quadro P5000显卡(16 GB GDDR5X显存),操作系统为Windows 10专业版。
表 2. 多尺度成像的真实与有效光学分辨率
| 尺度 | 物镜 | 真实分辨率 | 有效分辨率 | 数据量/毫米³ | ||
| 横向 | 轴向 | 横向 | 轴向 | |||
| 介观尺度 | 10×,NA0.3,水浸 | 1.2 μm | 17.8 μm | 0.8 μm | 11.8 μm | 5.5GB |
| 微观尺度 | 25×,NA 1.1水浸 | 0.3 μm | 1.1 μm | 0.2 μm | 0.8 μm | 500GB |
| 纳米尺度 | 25×,NA 1.1水浸 | 0.3 μm | 1.1 μm | 0.1 μm | 0.3 μm | 8TB |
¶ 参考文献
Adhya, D., Chennell, G., Crowe, J. A., Valencia-Alarcón, E. P., Seyforth, J., Hosny, N. A., Yasvoina, M. V., Forster, R., Baron-Cohen, S., Vernon, A. C. et al. (2021). Application of Airy beam light sheet microscopy to examine early neurodevelopmental structures in 3D hiPSC-derived human cortical spheroids. Mol. Autism 12, 4. doi:10.1186/s13229-021-00413-1
Albanese, A., Swaney, J. M., Yun, D. H., Evans, N. B., Antonucci, J. M., Velasco, S., Sohn, C. H., Arlotta, P., Gehrke, L. and Chung, K. (2020). Multiscale 3D phenotyping of human cerebral organoids. Sci. Rep. 10, 21487. doi:10.1038/ s41598-020-78130-7
Baumgart, E. and Kubitscheck, U. (2012). Scanned light sheet microscopy with confocal slit detection. Opt. Express 20, 21805-21814. doi:10.1364/OE.20. 021805
Benito-Kwiecinski, S., Giandomenico, S. L., Sutcliffe, M., Riis, E. S., Freire-Pritchett, P., Kelava, I., Wunderlich, S., Martin, U., Wray, G. A., McDole, K. et al. (2021). An early cell shape transition drives evolutionary expansion of the human forebrain. Cell 184, 2084-2102.e19. doi:10.1016/j.cell. 2021.02.050
Bershteyn, M., Nowakowski, T. J., Pollen, A. A., Di Lullo, E., Nene, A., Wynshaw-Boris, A. and Kriegstein, A. R. (2017). Human iPSC-derived cerebral organoids model cellular features of lissencephaly and reveal prolonged mitosis of outer radial glia. Cell Stem Cell 20, 435-449.e4. doi:10.1016/j.stem.2016.12.007
Bhaduri, A., Andrews, M. G., Mancia Leon, W., Jung, D., Shin, D., Allen, D., Jung, D., Schmunk, G., Haeussler, M., Salma, J. et al. (2020). Cell stress in cortical organoids impairs molecular subtype specification. Nature 578, 142-148. doi:10.1038/s41586-020-1962-0
Brémond Martin, C., Simon Chane, C., Clouchoux, C. and Histace, A. (2021). Recent trends and perspectives in cerebral organoids imaging and analysis. Front. Neurosci. 15, 629067. doi:10.3389/fnins.2021.629067
Burgers, J., Pavlova, I., Rodriguez-Gatica, J. E., Henneberger, C., Oeller, M., Ruland, J. A., Siebrasse, J. P., Kubitscheck, U. and Schwarz, M. K. (2019). Light-sheet fluorescence expansion microscopy: fast mapping of neural circuits at super resolution. Neurophoton 6, 015005. doi:10.1117/1.NPh.6.1.015005
Chen, B.-C., Legant, W. R., Wang, K., Shao, L., Milkie, D. E., Davidson, M. W., Janetopoulos, C., Wu, X. S., Hammer, J. A., Liu, Z. et al. (2014). Lattice lightsheet microscopy: imaging molecules to embryos at high spatiotemporal resolution. Science 346, 1257998. doi:10.1126/science.1257998
Chen, F., Tillberg, P. W. and Boyden, E. S. (2015). Expansion microscopy. Science 347, 543-548. doi:10.1126/science.1260088
Chozinski, T. J., Halpern, A. R., Okawa, H., Kim, H.-J., Tremel, G. J., Wong, R. O. L. and Vaughan, J. C. (2016). Expansion microscopy with conventional antibodies and fluorescent proteins. Nat. Methods 13, 485-488. doi:10.1038/nmeth.3833
Dean, K. M., Roudot, P., Welf, E. S., Danuser, G. and Fiolka, R. (2015). Deconvolution-free subcellular imaging with axially swept light sheet microscopy. Biophys. J. 108, 2807-2815. doi:10.1016/j.bpj.2015.05.013
Dekkers, J. F., Alieva, M., Wellens, L. M., Ariese, H. C. R., Jamieson, P. R., Vonk, A. M., Amatngalim, G. D., Hu, H., Oost, K. C., Snippert, H. J. G. et al. (2019). High-resolution 3D imaging of fixed and cleared organoids. Nat. Protoc. 14, 1756-1771. doi:10.1038/s41596-019-0160-8
Di Lullo, E. and Kriegstein, A. R. (2017). The use of brain organoids to investigate neural development and disease. Nat. Rev. Neurosci. 18, 573-584. doi:10.1038/ nrn.2017.107
Dodt, H-U., Leischner, U., Schierloh, A., Jahrling, N., Mauch, C. P., Deininger, K., Deussing, J. M., Eder, M., Zieglgansberger, W. and Becker, K. (2007). Ultramicroscopy: three-dimensional visualization of neuronal networks in the whole mouse brain. Nat. Methods 4, 331-336. doi:10.1038/ nmeth1036
Edwards, S.J., Carannante, V, Kunigk,K,Ring, H, Tararuk, T.,Halk, F, Blom,, nelt, .and Brimar, H. (200)High-esolution maging of t spheroids and organoids enabled by expansion microscopy. Front. Mol. Biosci. 7, 208. doi:10.3389/fmolb.2020.00208
Ellefsen, K. L. and Parker, I. (2018). Dynamic imaging with a simplified lattice light-sheet microscope: a sideways view of subcellular puffs. Cell Calcium 71, 34-44. doi:10.1016/j.ceca.2017.11.005
Fu, Q., Martin, B. L., Matus, D. Q. and Gao, L. (2016). Imaging multicellular specimens with real-time optimized tiling light-sheet selective plane illumination microscopy. Nat. Commun. 7, 11088. doi:10.1038/ncomms11088
Gao, R., Asano, S. M., Upadhyayula, S., Pisarev, I., Milkie, D. E., Liu, T.-L., Singh, V., Graves, A., Huynh, G. H., Zhao, Y. et al. (2019). Cortical column and whole-brain imaging with molecular contrast and nanoscale resolution. Science 363, eaau8302. doi:10.1126/science.aau8302
Giandomenico, S. L., Mierau, S. B., Gibbons, G. M., Wenger, L. M. D., Masullo, L., Sit, T., Sutcliffe, M., Boulanger, J., Tripodi, M., Derivery, E. et al. (2019). Cerebral organoids at the air-liquid interface generate diverse nerve tracts with functional output. Nat. Neurosci. 22, 669-679. doi:10.1038/s41593-019- 0350-2
Gonzalez, R. C., Woods, R. E. and Eddins, S. L. (2009). Digital Image Processing Using MATLAB. s.l: Gatesmark Publshing.
Huisken, J., Swoger, J., Del Bene, F., Wittbrodt, J. and Stelzer, E. H. (2004). Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science 305, 1007-1009. doi:10.1126/science.1100035
Iefremova, V., Manikakis, G., Krefft, O., Jabali, A., Weynans, K., Wilkens, R., Marsoner,F,Brand,B,Mller,J.,Kch,Pal. 2017)organoidad model of cortical development identifies non-cell-autonomous defects in Wnt signaling contributing to Miller-Dieker syndrome. Cell Rep. 19, 50-59. doi:10. 1016/j.celrep.2017.03.047
Ku, T., Swaney, J., Park, J.-Y., Albanese, A., Murray, E., Cho, J. H., Park, Y.-G., Mangena, V., Chen, J. and Chung, K. (2016). Multiplexed and scalable superresolution imaging of three-dimensional protein localization in size-adjustable tissues. Nat. Biotechnol. 34, 973-981. doi:10.1038/nbt.3641
LaMonica, B. E., Lui, J. H., Hansen, D. V. and Kriegstein, A. R. (2013). Mitotic spindle orientation predicts outer radial glial cell generation in human neocortex. Nat. Commun. 4, 1665. doi:10.1038/ncomms2647
Lancaster, M. A., Renner, M., Martin, C.-A., Wenzel, D., Bicknell, L. S., Hurles, M. E., Homfray, T., Penninger, J. M., Jackson, A. P. and Knoblich, J. A. (2013). Cerebral organoids model human brain development and microcephaly. Nature 501, 373-379. doi:10.1038/nature12517
Lancaster, M. A., Corsini, N. S., Wolfinger, S., Gustafson, E. H., Phillips, A. W., Burkard, T. R., Otani, T., Livesey, F. J. and Knoblich, J. A. (2017). Guided selforganization and cortical plate formation in human brain organoids. Nat. Biotechnol. 35, 659-666. doi:10.1038/nbt.3906
Masselink, W., Reumann, D., Murawala, P., Pasierbek, P., Taniguchi, Y., Bonnay, F., Meixner, K., Knoblich, J. A. and Tanaka, E. M. (2019). Broad applicability of a streamlined ethyl cinnamate-based clearing procedure. Development 146, dev166884. doi:10.1242/dev.166884
Neyra, E. G., Martı́nez-Matos, Ó. and Vaveliuk, P. (2020). Ultra-long light sheets via curved beam intercrossing. OSA Continuum 3, 2062-2069. doi:10.1364/ OSAC.393073
Pasca, A. M., Sloan, S. A., Clarke, L. E., Tian, Y., Makinson, C. D., Huber, N., Kim, C. H., Park, J.-Y., O’Rourke, N. A., Nguyen, K. D. et al. (2015). Functional cortical neurons and astrocytes from human pluripotent stem cells in 3D culture. Nat. Methods 12, 671-678. doi:10.1038/nmeth.3415
Peitz, M, Krutenko, T. and Brustle, O. (2020). Protocol for the standardized generation of forward programmed cryopreservable excitatory and inhibitory forebrain neurons. STAR Protoc. 1, 100038. doi:10.1016/j.xpro.2020.100038
Pollen, A. A., Bhaduri, A., Andrews, M. G., Nowakowski, T. J., Meyerson, O. S., Mostajo-Radji, M. A., Di Lullo, E., Alvarado, B., Bedolli, M., Dougherty, M. L. et al. (2019). Establishing cerebral organoids as models of human-specific brain evolution. Cell 176, 743-756.e17. doi:10.1016/j.cell.2019.01.017
Preibisch, S., Saalfeld, S. and Tomancak, P. (2009). Globally optimal stitching of tiled 3D microscopic image acquisitions. Bioinformatics 25, 1463-1465. doi:10. 1093/bioinformatics/btp184
Qian, K., Huang, C. T.-L., Huang, C.-L., Chen, H., Blackbourn, L. W., Chen, Y., Cao, J., Yao, L., Sauvey, C., Du, Z. et al. (2014). A simple and efficient system for regulating gene expression in human pluripotent stem cells and derivatives. Stem Cells 32, 1230-1238. doi:10.1002/stem.1653
Qian, X., Song, H. and Ming, G. (2019). Brain organoids: advances, applications and challenges. Development 146, dev166074. doi:10.1242/dev.166074
Quadrato, G., Brown, J. and Arlotta, P. (2016). The promises and challenges of human brain organoids as models of neuropsychiatric disease. Nat. Med. 22, 1220-1228. doi:10.1038/nm.4214
Quadrato, G., Nguyen, T., Macosko, E. Z., Sherwood, J. L., Min Yang, S., Berger, D. R., Maria, N., Scholvin, J., Goldman, M., Kinney, J. P. et al. (2017). Cell diversity and network dynamics in photosensitive human brain organoids. Nature 545, 48-53. doi:10.1038/nature22047
Schindelin, J., Arganda-Carreras, I., Frise, E., Kaynig, V., Longair, M., Pietzsch, T., Preibisch, S., Rueden, C., Saalfeld, S., Schmid, B. et al. (2012). Fiji: an open-source platform for biological-image analysis. Nat. Methods 9, 676-682. doi:10.1038/nmeth.2019
Schwarz, M. K. and Kubitscheck, U. (2021). Expansion light sheet fluorescence microscopy of extended biological samples: applications and perspectives. Prog. Biophys. Mol. Biol. 168, 33-36. doi:10.1016/j.pbiomolbio.2021.09.004
Silvestri, L., Bria, A., Sacconi, L., Iannello, G. and Pavone, F. S. (2012). Confocal light sheet microscopy: micron-scale neuroanatomy of the entire mouse brain. Opt. Express 20, 20582-20598. doi:10.1364/OE.20.020582
Stockhausen, A., Burgers, J., Rodriguez-Gatica, J. E., Schweihoff, J., Merkel, R., Prigge, J. M., Schwarz, M. K. and Kubitscheck, U. (2020). Hardwired lattice light-sheet microscopy for imaging of expanded samples. Opt. Express 28, 15587-15600. doi:10.1364/OE.393728
Tillberg, P. W., Chen, F., Piatkevich, K. D., Zhao, Y., Yu, C.-C., English, B. P., Gao, L., Martorell, A., Suk, H.-J., Yoshida, F. et al. (2016). Protein-retention expansion microscopy of cells and tissues labeled using standard fluorescent proteins and antibodies. Nat. Biotechnol. 34, 987-992. doi:10.1038/nbt.3625
Ueda, H. R., Erturk, A., Chung, K., Gradinaru, V., Chédotal, A., Tomancak, P. and Keller, P. J. (2020). Tissue clearing and its applications in neuroscience. Nat. Rev. Neurosci. 21, 61-79. doi:10.1038/s41583-019-0250-1
Velasco, S., Kedaigle, A. J., Simmons, S. K., Nash, A., Rocha, M., Quadrato, G., Paulsen, B., Nguyen, L., Adiconis, X., Regev, A. et al. (2019). Individual brain organoids reproducibly form cell diversity of the human cerebral cortex. Nature 570, 523-527. doi:10.1038/s41586-019-1289-x
Vettenburg, T., Dalgarno, H. I. C., Nylk, J., Coll-Lladó, C., Ferrier, D. E. K., Cimar, T., Gunn-Moore, F. J. and Dholakia, K. (2014). Light-sheet microscopy using an Airy beam. Nat. Methods 11, 541-544. doi:10.1038/nmeth. 2922
Yingling, J., Youn, Y. H., Darling, D., Toyo-Oka, K., Pramparo, T., Hirotsune, S. and Wynshaw-Boris, A. (2008). Neuroepithelial stem cell proliferation requires LIS1 for precise spindle orientation and symmetric division. Cell 132, 474-486. doi:10.1016/j.cell.2008.01.026
Zhao, S., Todorov, M. I., Cai, R., Maskari, R. A., Steinke, H., Kemter, E., Mai, H., Rong, Z., Warmer, M., Stanic, K. et al. (2020). Cellular and molecular probing of intact human organs. Cell 180, 796-812.e19. doi:10.1016/j.cell.2020.01.030