¶ 基于高密度多电极阵列和真实值验证的皮质兴奋性和抑制性神经元无监督识别流程
¶ Unsupervised pipeline for the identification of cortical excitatory and inhibitory neurons in high- density multielectrode arrays with ground-truth validation
原文下载:58010f2fdc7c879c2feec07517dad8ab2171.pdf
埃洛伊丝·吉罗1, 迈克尔·林恩2,3, 菲利普·文森特-拉马尔1,2, 让-克洛德·贝克2,3, 让-菲利普·蒂 维耶热1,2*
1.渥太华大学心理学院,加拿大渥太华;
2.渥太华大学神经动力学中心脑与心智研究所, 加拿大渥太华;
3渥太华大学医学院细胞与分子医学系,加拿大渥太华
¶ eLife 评估
在这篇手稿中,作者描述了一个软件包,用于对通过高密度微电极阵列获取的兴奋性和抑制性神经元产 生的动作电位进行自动微分。这项工作的价值在于它提供了一种工具,有望在体外自动识别这些神经元 类型。该工具具有扎实的基础,因为它提供了在高密度电极阵列上识别推定兴奋性和抑制性神经元的方 法,并可与其他现有的尖峰排序流程结合使用。
¶ 摘要
大规模细胞外记录技术推动了神经元回路的研究,但在扩展到对数千个神经元进行记录时,缺乏可靠识别细胞类型的方法。我们引入了 SpikeMAP,这是一种用于分析大规模体外皮层记录数据的流程,它将峰电位分类与利用病毒和光遗传学验证的细胞类型识别相结合。SpikeMAP 将数据分析与光遗传学、病毒学和药理学实验方案相结合,在对大量神经元进行记录的同时动态探测不同的细胞类型。该流程使用样条插值法拟合峰电位波形,以测量半幅值和峰峰值持续时间,应用主成分分析和 k 均值聚类来分离单个神经元信号,并使用线性判别分析来优化聚类的可分离性。通过时空峰电位波形特征确定通道信号源位置。将 SpikeMAP 应用于在 4096 通道阵列上记录的小鼠前额叶皮层切片数据时,它能够通过动作电位波形、法诺因子和空间互相关,有效区分规则放电的兴奋性神经元和快速放电的抑制性中间神经元。这个经过验证的工具包能够在高密度记录中全面描述不同细胞类型的神经元活动,为研究大脑中的微电路相互作用提供了一种可扩展的方法。
¶ 引言
高密度、大规模多电极阵列记录技术的出现,提升了我们对神经元群体如何表征和处理信息的理解,为 复杂网络动力学提供了关键见解(Riquelme等 人,2023;Hemberger等 人,2019;Shein-Idelson等 人,2 017;Thivierge等 人,2022)。此外,在体使用神经像素探针等硅记录探针,有助于揭示清醒行为小鼠 神经回路的基本原理(Lak等 人,2020;Peters等 人,2021)。然而,尽管人们日益认识到异质细胞类 型在回路水平计算中的重要性(Sylwestrak等 人,2022;Liu等 人,2021;Bugeon等 人,2022),仅根 据生理特性区分神经元细胞类型仍存在挑战。大规模电生理记录技术的可扩展性不仅取决于单个神经元 产生的动作电位的检测与分离(峰值排序),还取决于对不同细胞类型触发的动作电位进行可靠区分的 能力。
细胞外电位记录是研究神经活动的重要方法,具有高时空分辨率的优势,可同时监测数千个细胞的动 作电位(Magland等 ,2020;Buzsáki,2004)。基于细胞外动作电位波形的细胞类型分选已被广泛探索 。研究表明,兴奋性(E)与抑制性(I)神经元可根据动作电位波形进行区分(Hafizi等 ,2022;Ren等 ,2020;Barthó等 ,2004)。这类方法通常提取动作电位特征参数——如峰峰值时间差与半峰全宽—— 并将其与已知细胞类型的特征值进行匹配分类。该方法的理论基础源于细胞内记录研究:抑制性细胞( 如表达小清蛋白的GABA能中间神经元)具有短时程动作电位(Nowak等 ,2003;Peyrache与Destexhe, 2019;Kawaguchi与Kubota,1993;Connors与Gutnick,1990),而兴奋性锥体神经元则呈现长时程动作 电位(McCormick等 ,1985)。窄波形与宽波形动作电位的差异在前额叶皮层等多个新皮质区域中被证 实(Mitchell等 ,2007;Wilson等 ,1994;Constantinidis与Goldman-Rakic,2002;González-Burgos等 , 2005;Hasenstaub等 ,2005;Barthó等 ,2004;Tamura等 ,2004)。这种差异被推测源于宽放电锥体神 经元在动作电位后膜电位复极化过程更为缓慢(McCormick等 ,1985;Nowak等 ,2003;Hasenstaub等 ,2005)。在类似于负微分信号的细胞外记录中,细胞内膜电位,这使得在初始谷值之后出现一个更宽且更浅的峰值(Henze等 人,2000年)。
表1. 语言:编程语言,#电极:最大测试电极数,数据预处理:包含数据预处理(格式化、滤波、伪影去除),事件/通道定位:单事件定 位,波形提取:从波形中提取峰值特征(如峰峰值距离和半峰全宽),兴奋/抑制分类:兴奋性与抑制性细胞分类,验证:细胞类型分类 验证(+)=通过细胞内记录进行真实值验证但不包含兴奋/抑制验证,实验协议:集成实验协议以支持网络操控,无监督:无需人工干预 ,源代码可获取:全部代码开源可用。
| 方法 | 语言 | 电极数 | 数据预处理 | 事件/通道定位 | 波形提取 | 兴奋/抑制分类 | 验证 | 实验方案 | 源代码无监督可用 |
|---|---|---|---|---|---|---|---|---|---|
| spikeMAP | Python 和 Matlab | 4096 | + | + | + | + | + | + | + |
| Kilosort4 (Pachitariu 等, 2023) | Python | + | + | - | - | - | - | + | |
| SpyKING CIRCUS (Yger 等, 2018; Yger 等, 2023) | Python | 4225 | - | + | + | - | (+) | - | + |
| MountainSort (Chung 等, 2017) | Matlab 和 C++ | 16 | + | + | - | - | - | - | + |
| Herding Spikes (Hilgen 等, 2017) | Python | 4096 | + | + | + | - | (+) | + | + |
| YASS (Lee 等, 2017a; Lee 等, 2017b) | Python | 512 | - | + | - | - | - | - | + |
| JRCLUST (Jun, 2017, Jun, 2017) | Matlab 和 CUDA | 120 (1000) | + | + | - | - | (+) | - | + |
现代细胞外记录技术使研究人员能够监测大型、完整神经元网络的同步活动。然而,在密集样本中,单个细胞的活动可能被多个电极捕获,而每个电极又可能记录到多个邻近细胞的活动。因此,为分离单个细胞的信号,必须采用有效的尖峰分类技术。该技术通常包括过滤原始细胞外信号、通过阈值检测动作电位、提取尖峰特征,以及通过聚类分析区分不同神经元(Rey等人,2015)。
目前已开发出多种各具特色的方法,包括Kilosort4(Pachitariu等人 ,2023)、SpyKING CIRCUS(Yger等人 ,2018)、MountainSort(Chung等人 ,2017)、Herding Spikes(Hilgen等人 ,2017)、YASS(Lee等人 ,2017a)和JRCLUST(Jun等人 ,2017)。表 1详细比较了这些方法。这些方法普遍缺乏以生物学验证方式识别细胞类型的能力。尽管有证据表明不同细胞类型的动作电位动力学存在差异,且光遗传学技术已被应用(Hilgen等人 ,2017),但据我们所知,目前尚无大规模验证研究证明细胞外波形可可靠区分细胞类型。利用细胞类型间遗传差异的方法可提供预测细胞类型的统计指标,如法诺因子(Becchetti等人 ,2012)。这些方法使研究人员能够通过事后分析推断最可能被采样的细胞类型。然而,目前仍需开发完整集成方案:将开源数据分析流程与经过验证的实验方案相结合,实现在记录过程中进行高密度细胞类型特异性激活。这种方法将能以灵活、可扩展且普适的方式研究密集复杂的神经回路。
在此,我们推出spikeMAP——一种开源、无监督且可扩展的峰电位排序分析流程,该流程经过高密度微电极阵列验证可实现细胞类型分类。关键创新在于,我们通过采用稳定阶跃函数视蛋白的实验方案,将分析与动态探测不同神经元群体深度整合,在验证排序效果的同时规避了CMOS芯片设计常见的光诱导伪影问题(Fiscella等 人,2012)。该分析流程利用密集采样优势,通过估算每个检测到的峰电位的胞体位置,生成与单个神经元对应的清晰分离聚类簇。我们采用信号插值技术提取峰电位波形特征,以此估算半幅值与峰峰值持续时间,继而通过主成分分析与k均值聚类识别阵列中单通道的非相关信号。采用线性判别分析评估聚类的最优分离度,并基于峰电位波形在阵列中的时空特征定位各通道信号源。进一
步根据波形特性将细胞分类为推定兴奋性或抑制性神经元。最后,我们开发了一种利用长时程光遗传学(阶跃函数视蛋白)的新方法,在小鼠前额叶皮层急性切片中实现细胞类型特异性刺激,为统计方法提供了生物学验证。spikeMAP整体而言一个供实验人员使用的综合性工具包,可灵活探究全脑各种神经回路中的细胞类型特异性计算。
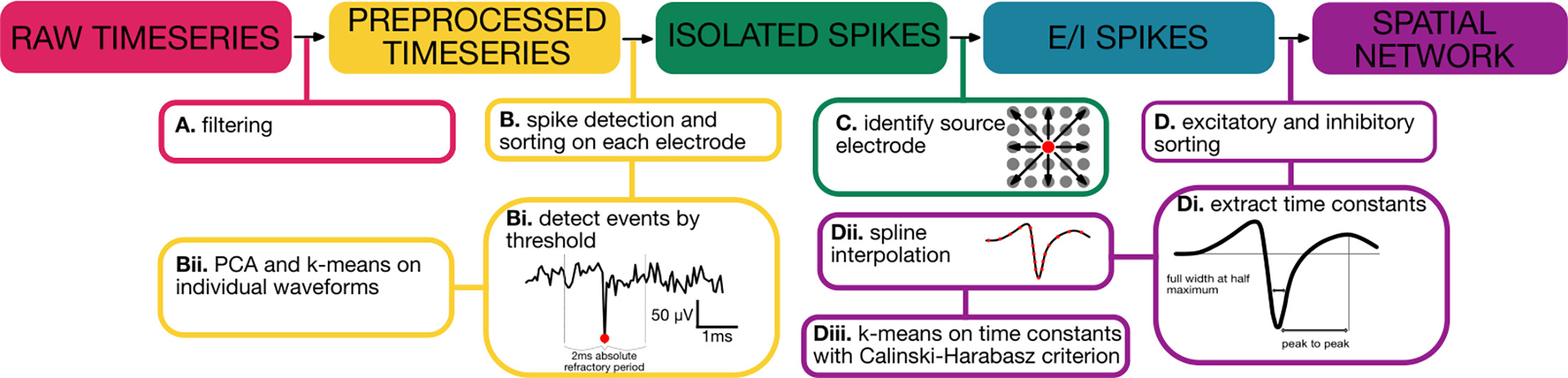
图1. 所提出方法的工作流程图。(A) 通过带通滤波(300-5000 Hz)实现数据滤波。尖峰检测在滤波后的电压信号上进行,但每个尖峰周围的原始电压被用于避免信号失真。(B) 各电极上的尖峰排序。(Bi) 使用阈值法(滤波信号均值的6倍标准差)检测事件。(Bii) 对单个波形进行主成分分析和k均值聚类(k=2)。© 阵列上精确定位胞体位置。(D) 兴奋性与抑制性分类。(Di) 通过半峰全宽和峰峰值距离提取尖峰时间常数动力学特征。(Dii) 采用插值电压提高分辨率。(Diii) 基于波形动力学特征通过k均值聚类区分细胞类型。
¶ 结果
¶ 脉冲排序
我们的峰电位分类流程(图 1)主要执行三项任务:(i)检测与假定体细胞峰电位相关的电压偏转,(ii)通过峰电位聚类识别单个假定神经元,以及(iii)在高密度记录中实现单细胞的空间定位。我们通过对小鼠内侧前额叶皮层进行定向高密度记录来演示该流程的应用(图 2A;参见材料与方法)。首先,通过阈值穿越法检测假定峰电位出现时间(图 2Bi;Lewicki, 1998),从滤波信号均值中识别负信号峰值(参见材料与方法)。最优阈值选择是区分峰电位与噪声的关键,因为每个电极检测到的峰电位数量高度依赖于所设阈值(图 2C)。本研究通过将各通道的电压直方图拟合为高斯分布,从而分离峰电位活动与正态分布噪声,以此确定最佳检测阈值(图 2D)。当阈值为–3σ时,信号主要可归因于高斯噪声,而信号与噪声的分离约在–4σ阈值时开始显现。为避免同一事件的重复检测,我们设置了2毫秒的绝对不应期以剔除 <内的连续事件。尽管滤波对事件检测至关重要,但可能引起信号失真(Yael和Bar-Gad, 2017)。为解决该问题,我们提取了每个峰电位时刻未经滤波的原始电压(图 2Bii),从而为每个通道生成一系列高频(18 kHz)波形。
在高密度制备中,单个电极可记录多个细胞的活动。为解决该问题,我们对各通道识别出的尖峰波形进行主成分分析(Rossant等 人,2016),结果显示存在推测对应单个细胞的明显聚类(图 2E),表明某些通道记录的电压信号可能来自多个神经元。SpikeMAP套件还提供例行程序,可在单个通道周围选择半径范围,从而将相邻通道组纳入主成分分析。随后采用k均值聚类分析法从主成分中提取聚类。为评估该方法的数据分离效果,我们对标记的主成分进行线性判别分析(Hill等 人,2011),使分析方法始终保持完全无监督状态。在SpikeMAP中,k均值聚类的最佳数量可通过Calinski-Harabasz准则(Calinski与Harabasz,1974)选择或由用户预先指定。
为确保每个单细胞的活动仅被记录一次,我们在检测到特定细胞的电极周围设置了250 μm的半径范围。该半径对应于单个神经元胞体周围细胞外电场的近似扩散范围(Jia等 人,2019)。在此半径内,我们识别出平均波形中具有最大负向偏转的通道。先前研究观察到,细胞外信号的最大负幅值源自轴突起始段(AIS;Bakkum等 人,2019;Obien等 人,2014)。利用AIS的空间定位特性,呈现最大负向偏转的通道很可能最接近特定细胞的推定胞体(Jia等 人,2019)。该通道的活动与胞体活动一致,显示峰值电压随远离推定胞体而衰减(图 2H;Delgado Ruz与Schultz,2014;Pettersen与Einevoll,2008;Lindén等人,2011;Mechler等 人,2011;图 3)。相邻电极的可视化结果显示电压活动具有复杂结构,可能源于单个神经元不同区室内的活动传播(图 2F;Shein-Idelson等 人,2017;Obien等 人,2014)。作为额外验证步骤,SpikeMAP可计算用户定义半径内推定神经元之间的峰电位计数相关性。超过设定相关性阈值的信号可被排除,因为它们可能反映同一底层细胞。为进一步验证位于同一电极附近的单细胞可被区分,我们通过质心分析估算了相邻记录电极间的精确胞体位置(参见材料与方法)。这揭示了位置的细微变化,体现为各邻近电极上的不同权重(图 2I)。尽管存在这些细微变化,由于棘波定位的变化远小于电极间距,胞体位置仍可被归属至同一电极。
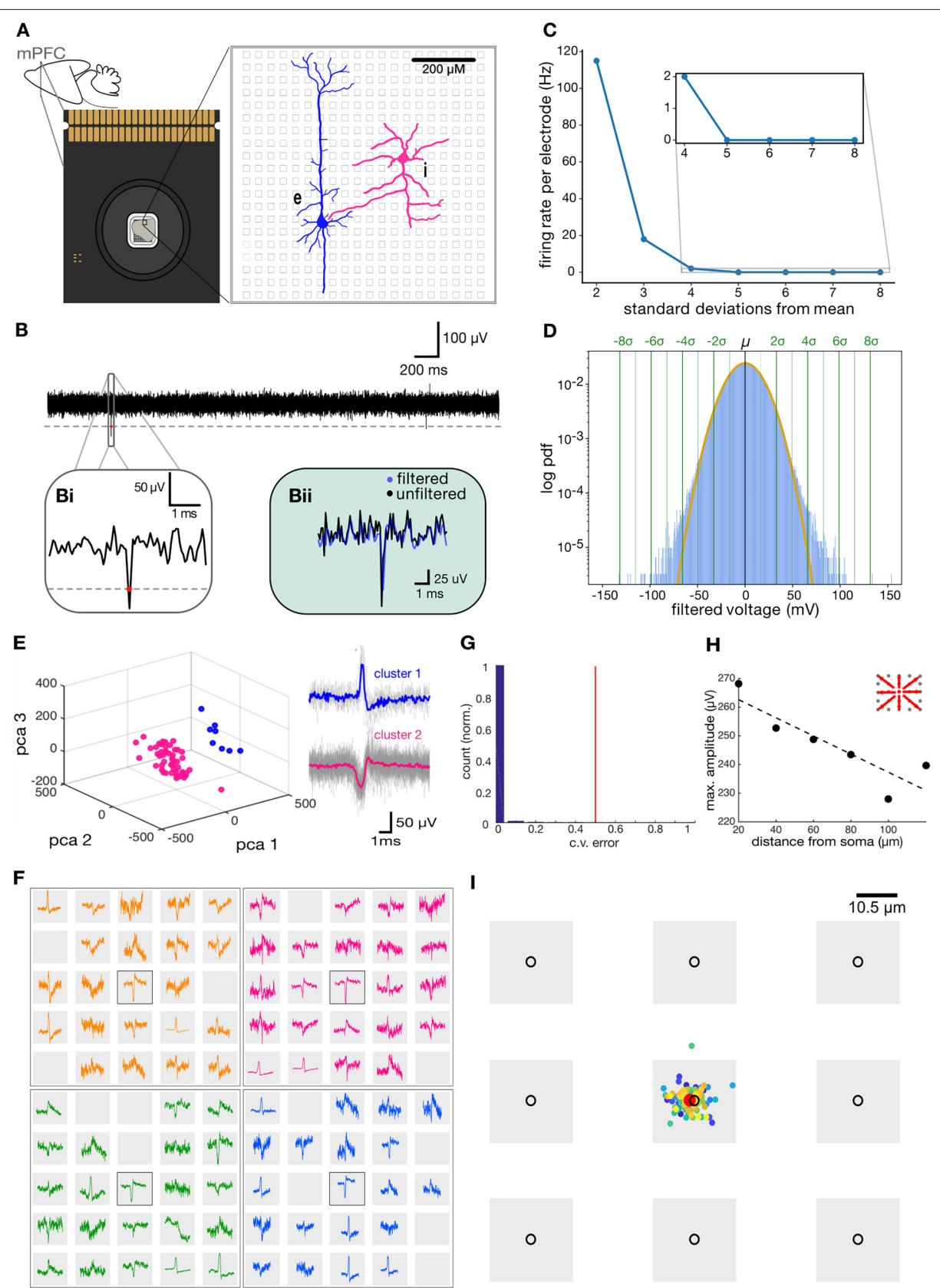
图2. 单电极动作电位的分离流程。(A) 多电极阵列记录装置示意图。(B) 单个电极的细胞外电压信号。(Bi)滤波电压信号上的锋电位检测。(Bii)检测到锋电位周围的原始电压信号提取。© 单电极放电频率与阈值选择的相关性。插图显示选定阈值(4倍标准差)下的放电频率值。(D)通过阈值设定从正态分布噪声中分离细胞活动信号。(E) 单电极上的锋电位分类。(F) 单个神经元产生的电流汇信号可覆盖多个电极(图中展示了四个不同神经元的。(G) 以不同颜色显示的单次记录中所有电极的十倍交叉验证直方分布图,其中红色表示随机性能水平。(H) 电压振幅随与最高电极峰值距离变化的函数关系,插图展示了从中心电极测量的方向。(I) 单个神经元胞体所有峰电位的质心分布位置,红色标记表示均值点。
总之,SpikeMAP提供了一个端到端的流程,可在高密度多电极阵列上执行峰值排序。该流程的某些要素与相关方法类似(表 1),包括使用电压滤波、PCA和k均值聚类。其他要素则具有创新性,包括使用样条插值、线性判别分析以及识别推测的兴奋性和抑制性细胞的能力。在流程的不同步骤中,可通过以下方式定制剔除峰值的条件:(1)对滤波电压设置严格阈值;(2)设定电压信噪比的最小截止值(图 4);(3)采用线性判别分析评估簇间分离度;(4)为推测的神经元设置最低峰值频率;(5)通过哈蒂根统计凹陷检验检测峰值爆发;(6)电压随远离推测胞体而衰减;(7)相邻通道间设置最大峰值计数相关性。这些标准共同使SpikeMAP用户能够精确控制与自动峰值排序相关的参数。
¶ 基于动作电位波形动力学的细胞类型分类
尽管技术不断进步,仅依靠细胞外电压来识别特定细胞类型的能力仍然具有挑战性。当前的数据处理方法通常依赖波形动力学(Barthó等 人,2004)来对参与反应的兴奋性和抑制性单元进行分类。随着技术发展持续推动高密度阵列的实现,波形尖峰分析对邻近单元信号产生的噪声日益敏感。本研究首先基于现有细胞类型特异性波形动力学知识,结合样条插值法实施分类方法,以在密集网络中区分假定的兴奋性与抑制性神经元。随后我们通过联合应用病毒策略、光遗传学及药理干预手段,同时记录受刺激小鼠前额叶皮层网络的信号,对该分类方法进行了验证。
为了更精确地描述每个尖峰信号的形态,我们首先采用分段三次样条插值法对每个波形进行拟合。这种方法允许在已知数据点边界内进行统计推断。尽管存在多种插值基函数可选,但三次样条能在已知数据点间实现平滑插值,并具有较高的曲线拟合精度(Ramsay和Silverman,1997)。通过设定90 kHz的查询点,我们计算了每个推定细胞的平均拟合波形(Blanche与Swindale,2006,详见材料与方法)。虽然90 kHz的分辨率已能对尖峰波形进行合理估计,该数值在SpikeMAP中可作为可调参数进行修改。
接下来,我们利用从胞体位置平均锋电位波形中获取的两个时间常数来识别假定的兴奋性神经元和抑制性神经元:(1) 锋电位在半最大振幅处的全宽(FW),以及(2) 峰峰值(PP)持续时间(图 5A)(Sirota等 人,2008;Sakata和Harris,2009;Barthó等 人,2004;Insel和Barnes,2015)。尽管有研究对基于锋电位宽度的分类准确性提出质疑(Moore和Wehr,2013),但许多研究表明该方法具有良好的分类效能(Nowak等 人,2003;Kawaguchi和Kubota,1997;Barthó等 人,2004。Madisen等人,2012;Stark等人,2013;Cardin等人,2009;Cohen与Mizrahi,2015)。对全部同步记录的体细胞电极获取的FW和PP值进行K均值聚类,并通过Calinski-Harabasz准则确定最优聚类数量(Calinski与Harabasz,1974;Vendramin等人,2009)。为验证目的,在流程各环节采用严格标准,导致本文报告的神经元数量较少。根据FW和PP值识别出对应推定兴奋性(E)与抑制性(I)神经元的聚类(图5A)。I细胞被识别为具有快速FW和PP时间常数的单一聚类(Barthó等人,2004)。我们提取了MEA上兴奋性与抑制性神经元的推定胞体位置(图5B),并通过点阵图展示其放电活动(图5C)(放电率分布见图6)。推定I细胞的空间位置可能反映了内侧前额叶皮层视蛋白的注射位点。推定I细胞的其他特性与既往研究一致,包括较高的Fano因子(图5D;Becchetti等人,2012)及快速的空间配对相关性衰减(图5E;Peyrache等人,2012)。此外,SpikeMAP包含对单个细胞峰电位间隔分布执行哈蒂根统计双峰检验的流程,以检测推定爆发性神经元。这些结果将既往发现扩展至大规模记录,但仍需通过真实验证评估该方法在多大程度上可成为生物学身份的合理代理指标,如下文所述。
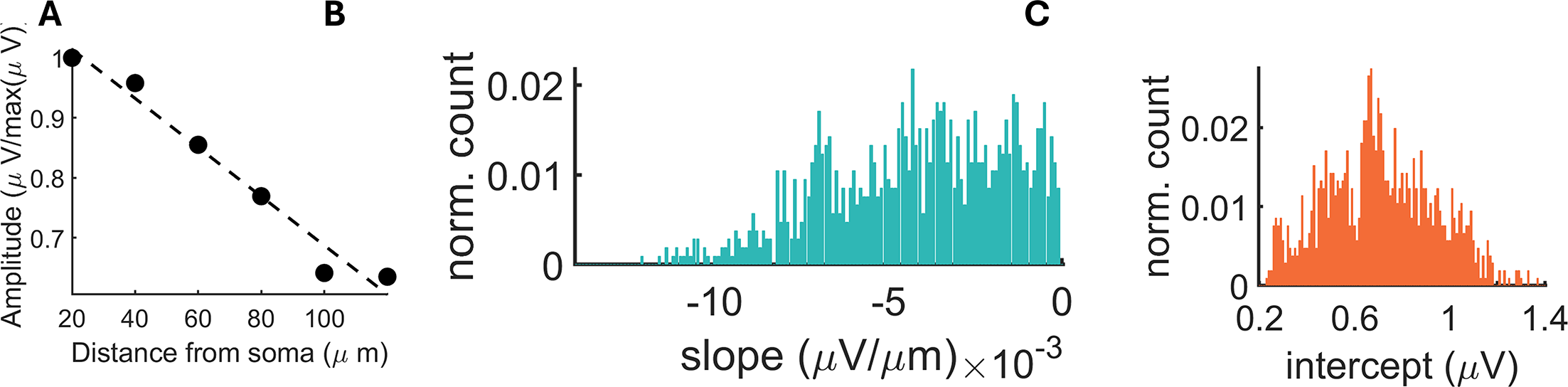
图3. 电压随推定胞体距离增加而衰减的趋势。(A) 电压幅度随最高电极峰值点距离变化的函数关系。本次分析排除了电压随电极峰值点距离增加而升高的推定胞体。(B—C) 单次记录中所有推定胞体(样本量=1950)的斜率和截距分布情况。
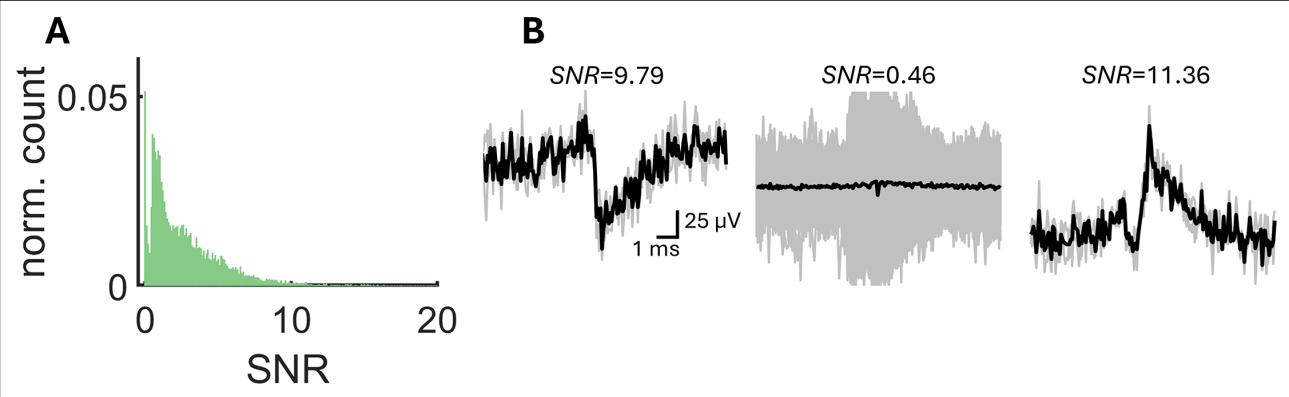
图4. 单通道信噪比分析。 (A) 单个记录的所有尖峰排序集群的信噪比分布(N=8059)。 (B) 围绕单个尖峰的平均电压(黑线)和个体电压(灰线)示例。
¶ 用于细胞分类的阶梯函数视蛋白验证方法
尽管多电极阵列已能同时记录数千个电极信号,但其对光敏互补金属氧化物半导体(CMOS)电路的依赖限制了研究人员将其与光遗传学技术结合使用的能力。由于快速扰动大型网络中不同神经元群体的能力可为局部回路如何表征信息提供重要见解(Lemon等 人,2021;Buzsáki,2010),因此验证能够在多电极阵列中对特定细胞群体进行光遗传学操控的方法具有广泛的研究价值。
我们验证了一种采用稳定阶跃函数视蛋白(双稳态神经状态开关;Berndt等 人,2009)的实验方案。该视蛋白可通过蓝光短时刺激以阶跃方式持续激活数分钟,并能被绿黄光灭活。这种病毒与光遗传学策略结合基于CMOS的多电极阵列使用,可在初始光致伪影之外监测诱导产生的锋电位(图 7)。我们通过选择性诱导目标PV抑制性神经元产生锋电位验证了该方案——由于超过90%的此类神经元已被报道为快放电类型(Stringer等 人,2016),借此可验证先前被识别为推定抑制性中间神经元的细胞是否确实对应光激活细胞。
作为一项必要的验证步骤,我们向小鼠内侧前额叶皮层注射了表达阶梯函数视蛋白的病毒载体pAAVEf1a-DIO hChR2(C128S/D156A)-EYFP。在等待2-3 周转染期后,通过体外脑片制备技术对定位在PV中间神经元附近锥体神经元进行全细胞记录(图 8A;详见材料与方法)。蓝光光刺激可有效激活PV中间神经元,表现为光诱导抑制性突触后电流稳定、快速且一致地出现(从锥体神经元记录到的IPSCs)(注意这些细胞没有直接光电流;图 8B)。随后施加的短脉冲黄光降低了锥体神经元上的IPSCs频率,表明阶跃函数视蛋白可通过阶段性光刺激以阶梯式被可靠地激活和失活(图8B)。在PV光激活后,被记录的锥体神经元持续接收到10-30 pA的高频抑制性突触后电流簇,随后振幅逐渐减小并伴随持续增强的抑制性输入速率,直至绿黄光使其失活,这反映了阶跃函数视蛋白的某种脱敏现象(图 8B)。
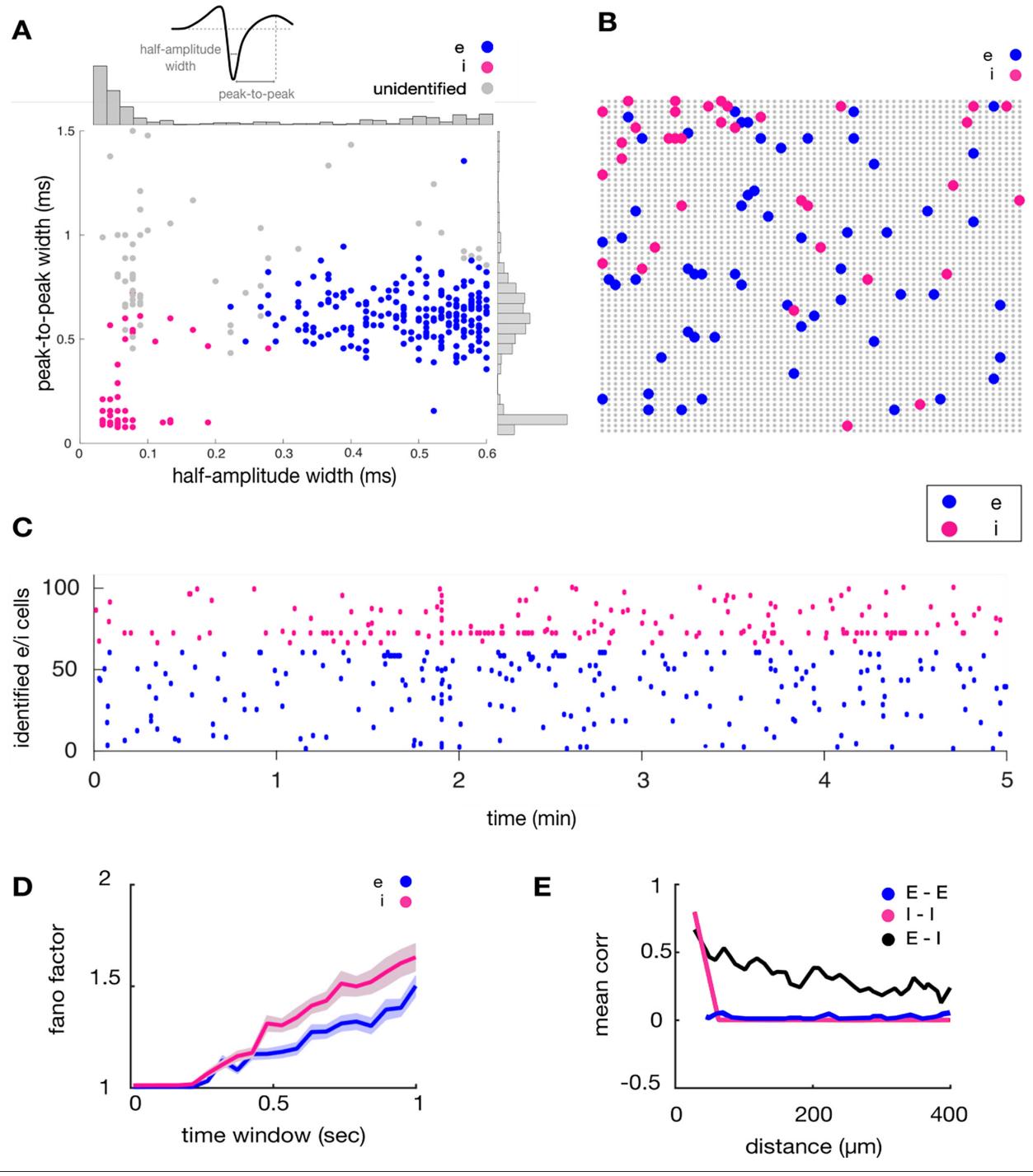
图5. 推定兴奋性与抑制性单元的识别。(A) 兴奋性与抑制性细胞呈现不同的波形动力学特征 (B) 对应细胞定位分布 © 兴奋性与抑制性神经元活动的点阵图 (D) 推定兴奋性与抑制性细胞的法诺因子。阴影区域表示标准误。横轴显示用于计算法诺因子的递增时长时序区间 (E) 电极对间平均互相关函数随空间距离的变化关系。采用100毫秒非重叠时间窗计算相关性,最终结果按所有神经元对的空间距离进行平均值统计。
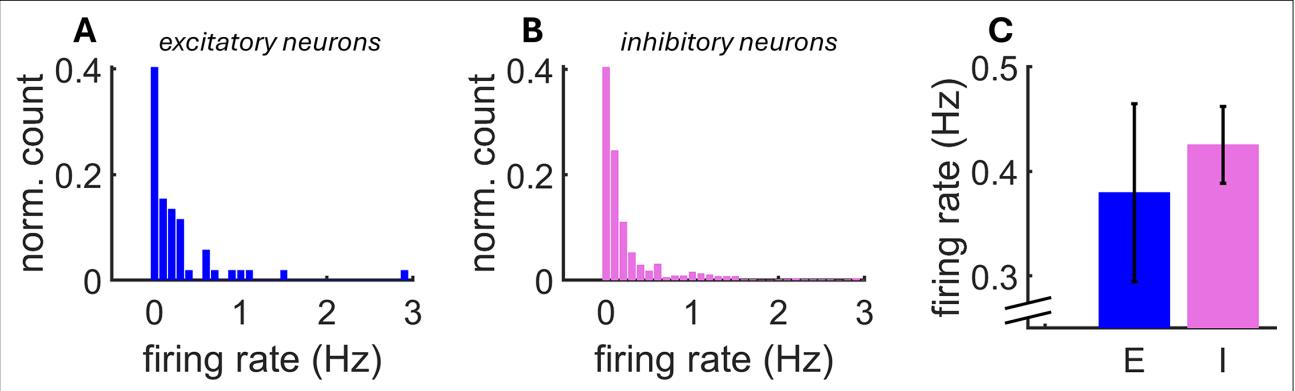
图6. 在推定的兴奋性神经元 (A) (N=570) 和抑制性神经元 (B) (N=54) 群体中平均放电率的分布。 © 神经元间的平均放电率。 垂直线条:标准误。
为了表征阶跃函数视蛋白的不应期,我们以不同试次间隔施加重复光刺激,并测量稳态激活水平(图8C-E)。较长的试次间隔(≥120 秒;图 8E)使表达细胞得以恢复,避免了20 秒和60 秒试次间隔下观察到的稳态激活降低(图 8C-D)。具体而言,20 秒、60 秒和120 秒间隔对应的平均IPSCs/秒分别为12.64±0.27、17.32±0.15和26.29±0.31。采用Wilcoxon秩和检验对PSTH非参数数据进行统计比较发现:20 秒与120 秒条件之间存在显著差异(p=1.29×10⁻⁶,n=4500 与3000次试验),60 秒与120 秒条件之间亦存在显著差异(p=0.032,各条件n=3000次试验)。各条件实际试验次数为:20 秒间隔5次、60 秒间隔6次、120 秒间隔6次。
此外,在较长间隔试验中,视蛋白失活导致的放电频率降低更为显著(图 8E)。这些结果为多电极阵列记录提供了关键见解。该协议允许我们使用与多电极阵列技术兼容的工具,以时间精准的方式选择性地激活和抑制PV中间神经元。
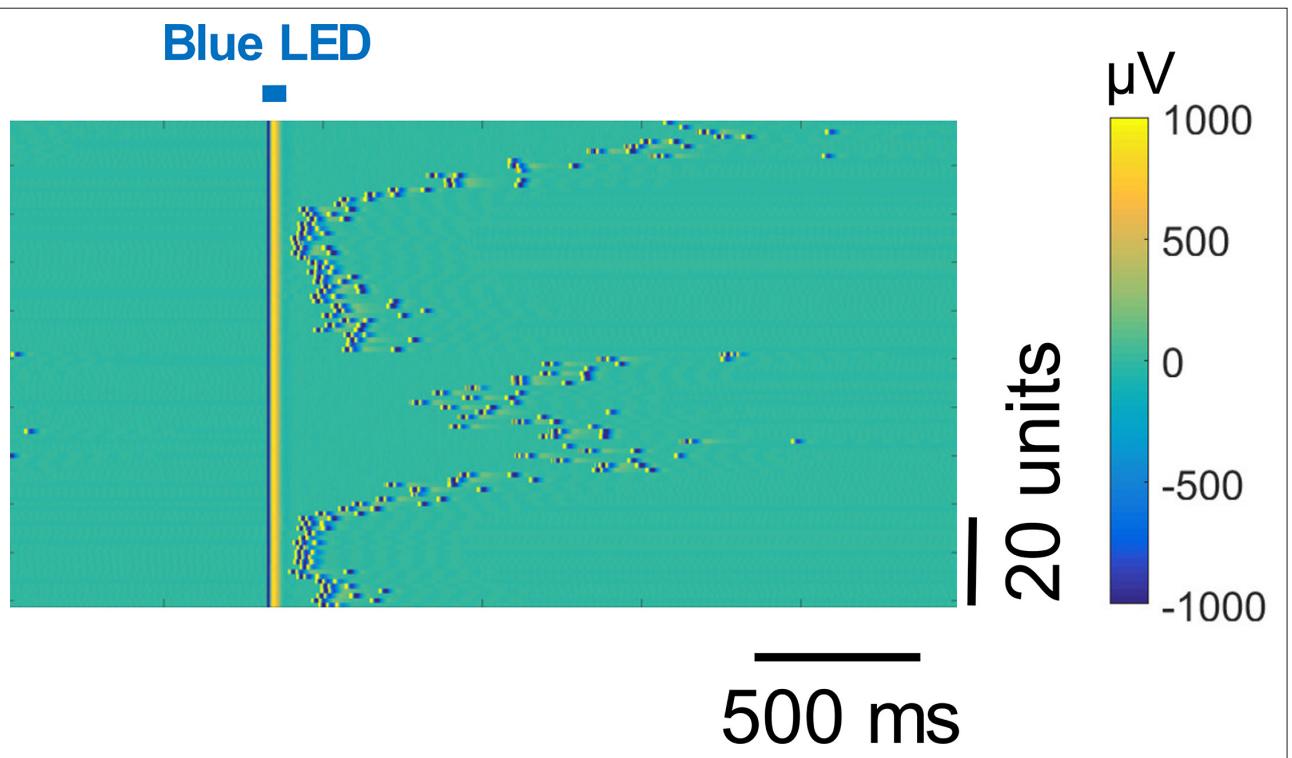
图7. 小鼠前额叶皮层记录中的光学伪影表明,施加短暂(1毫秒)的光脉冲会导致刺激后持续约5毫秒的饱和现象。
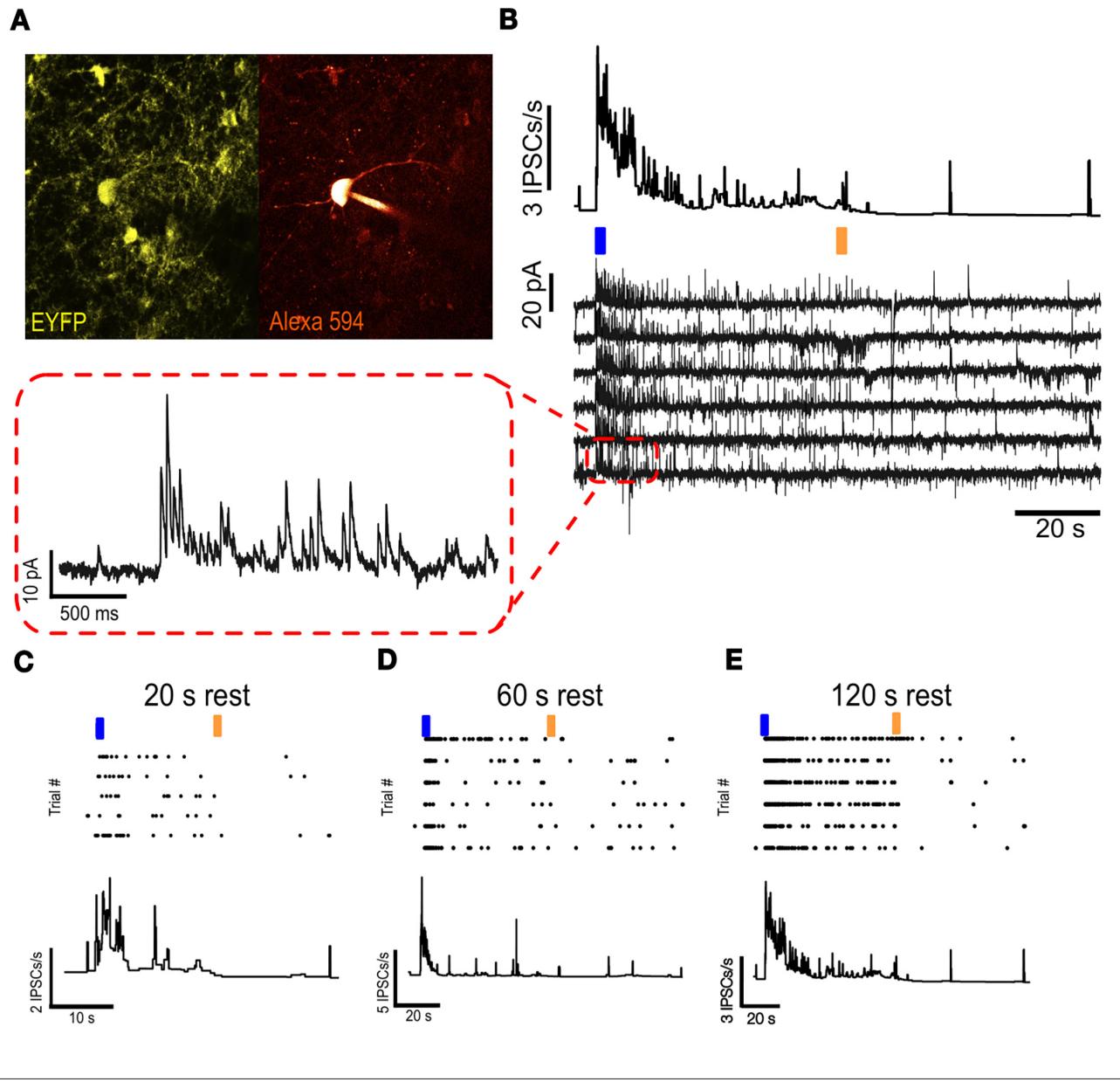
图8. 阶梯函数视蛋白的全细胞记录验证。(A) 视觉识别的PV神经元 (B) 光激活PV后接收抑制性电流的邻近锥体神经元 © 锥体细胞上记录的IPSCs,试验间隔20 秒 (D) 锥体细胞上记录的IPSCs,试验间隔60 秒 (E) 锥体细胞上记录的IPSCs,试验间隔120秒
¶ 多电极阵列上E/I分类的光遗传学验证
为验证先前基于波形的细胞类型分类方法在小鼠前额叶皮层切片中的有效性,我们在进行多电极记录的同时,使用阶跃函数视蛋白构建体选择性激活转染的PV中间神经元(表 2)。为防止光遗传激活在整个网络中传播,我们首先在10 μμM氰奎沙林(CNQX)和100 μμM苦味毒素(PTX)存在下进行记录,以分别阻断AMPA受体和GABAA受体(导致放电频率较基线降低0.0937 Hz,p<0.001)。我们采用100 毫秒蓝光脉冲进行光刺激,并检测所有已识别神经元的刺激后反应(n=8189,3个切片,来自3 只小鼠;图 9A)。在突触阻断剂存在的情况下,对目标PV中间神经元的光遗传激活使得被归类为该细胞群体的平均放电频率显著增加。
表2. SpikeMAP开源数据概览。数据集采集自四只小鼠,每套数据均执行一次光遗传刺激方案(详见材料与方法部分)。数据来源:https://doi.org/10.6084/m9.figshare.29416472。
| 文件名 | 动物编号 | 方案编号 | 识别出的兴奋性细胞数 | 识别出的抑制性细胞数 | 总记录时间(分钟) |
|---|---|---|---|---|---|
| R20211221_Slice1_Vars3.mat | 1 | 1 | 3687 | 47 | 8 |
| R20211213_Slice2_Vars3.mat | 2 | 1 | 2943 | 350 | 8 |
| R20211209_Slice3_Vars3.mat | 3 | 1 | 1994 | 712 | 8 |
| R20211219_Slice3_Vars3.mat | 4 | 2 | 1138 | 32 | 8 |
根据波形特性被归类为抑制性神经元(I神经元;图 9B,n=430,p<0.003)。而根据波形动力学被归类为兴奋性神经元的神经元,其平均放电率仅出现轻微下降(图 9B,n=7759,p<0.001)。对推定兴奋性和抑制性神经元锋电位波形特性的聚类分析为分类提供了进一步证据(图 10)。切片记录中的总体放电率相对较低,因此无法通过交叉相关性验证推定的兴奋性和抑制性神经元。虽然我们的药理学操作试图基本消除网络效应,但轻微的假定兴奋性神经元放电频率的降低可能反映了GABAB受体的参与-介导抑制(Kohl and Paulsen,2010)。
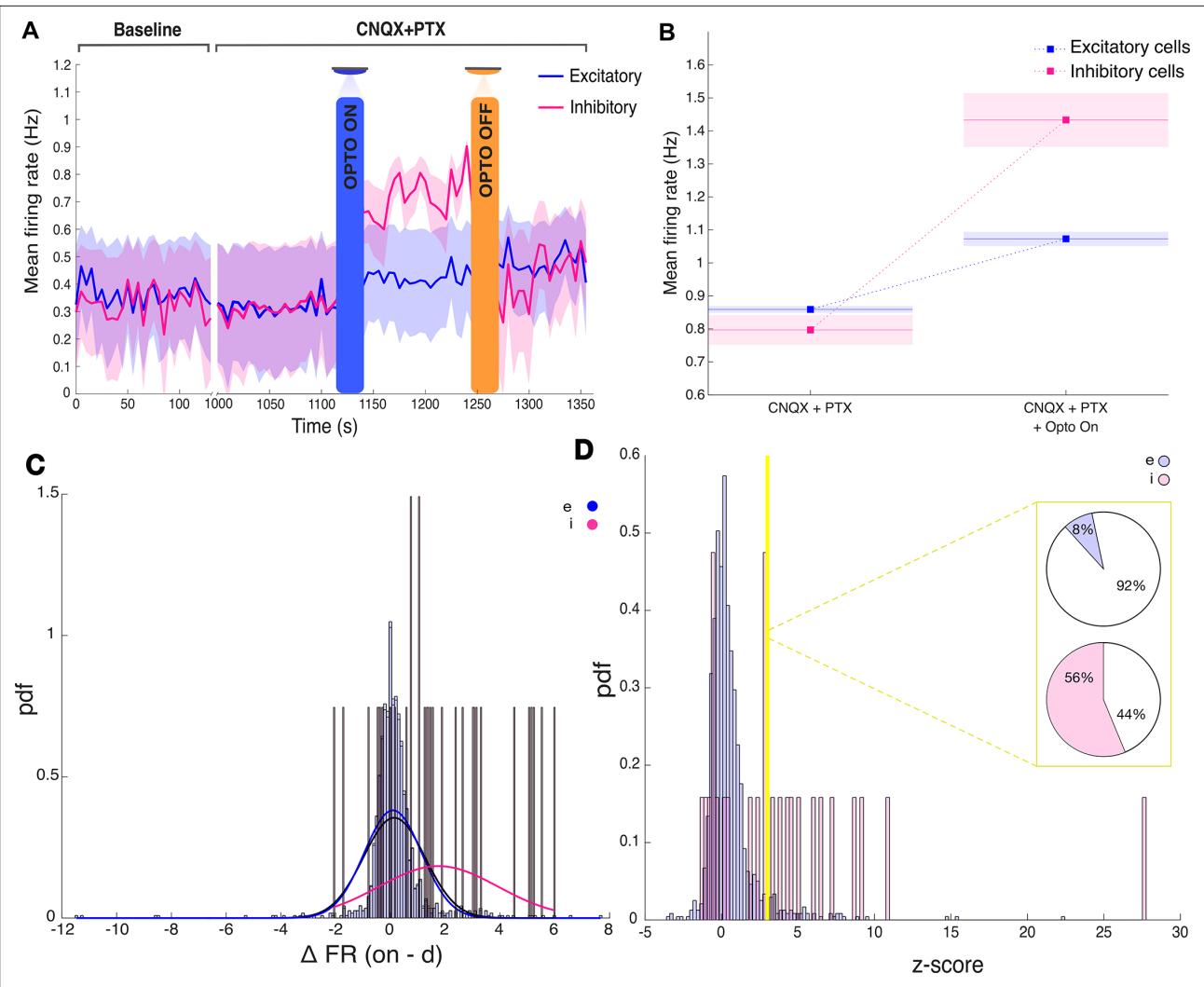
图9. 用于峰值分类的光遗传学抑制性细胞靶向 (A) 假定兴奋性与抑制性神经元的平均放电频率。阴影区域表示标准误差均值。(B) 光遗传学刺激后放电频率变化的量化分析。平均放电频率数据来自三只 小鼠的四组记录。© 放电频率的增量变化(药物干预期间)(D) 光遗传刺激引发的放电频率变化。
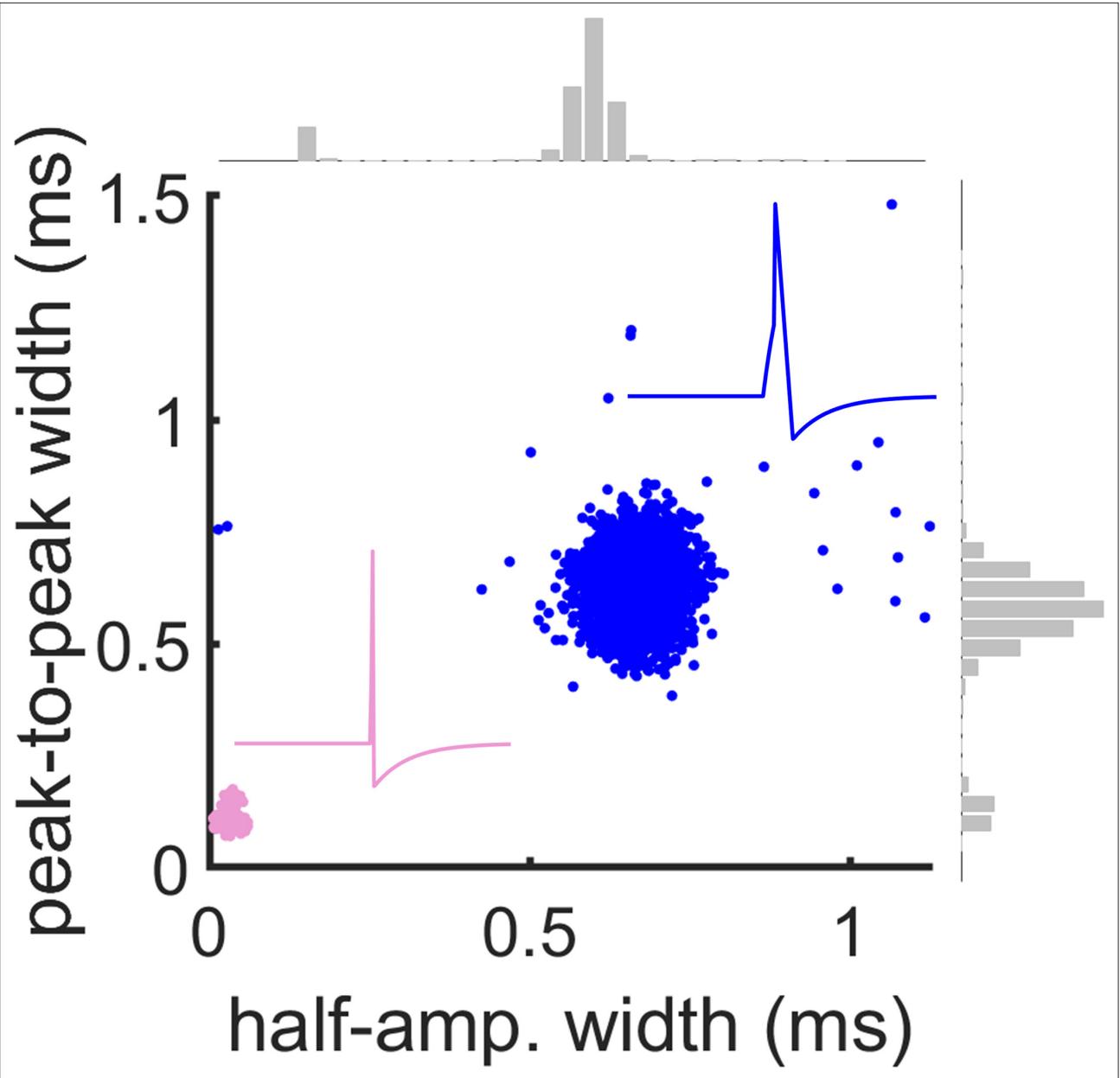
图10展示了光遗传学方案基线窗口中获取的兴奋性与抑制性神经元波形动态。插图为单个动作电位的示意图,分别呈现较宽的兴奋性神经元波形 与较窄的抑制性神经元波形 。
接下来,我们进一步详细考察了单个神经元对光刺激的反应模式,以便在单细胞水平验证我们的算法。在未使用突触阻断剂的情况下,个别神经元对光刺激表现出多样化的反应类型:包括放电频率无显著变化的神经元(ΔFR~0)、放电频率增加的神经元(ΔFR>0)以及放电频率降低的神经元(ΔFR<0;图9C)。我们采用严格标准来筛选那些在统计学上具有显著放电频率增加的神经元(参见材料与方法)。研究发现,在被归类为抑制性神经元的细胞中,56%表现出光诱导的放电频率提升。相反,在被归类为兴奋性的神经元中,仅8%在光刺激后出现放电频率增加。抑制性细胞对光响应的比例可能受到我们病毒策略的影响——该策略仅靶向部分表达小清蛋白的抑制性中间神经元,这些细胞主要分布在前边缘皮层和下边缘皮层亚区。此外,某些非小清蛋白抑制性中间神经元及部分兴奋性神经元可能因其波形特征被初步归类为抑制性神经元。
然而,实验明确识别出基因鉴定的PV神经元中的一个子集:那些在光刺激后表现出瞬态放电频率增加的神经元。通过与半峰全宽和峰峰值特征的比较,这些结果表明阶跃函数视蛋白方法是一种可行且可扩展的技术,可用于验证多电极阵列上的棘波波形分类。
综上所述,这为基于spikeMAP提取的波形特性对兴奋性和抑制性神经元进行细胞分类提供了证据,表明被识别为抑制性的细胞在光遗传学特异性反应中表现出变化。
¶ 讨论
细胞外电生理记录使得同时监测数千个神经元的放电活动成为可能。为有效解析这些动态信号,峰值排序成为记录分析的关键步骤。对基础神经活动的错误解读会严重影响网络动力学分析(Rey等 人,2015;Carlson与Carin,2019),这凸显了优化方法的必要性。随着技术发展不断推动高密度阵列的应用,鉴于数据规模已无法进行人工校审,将峰值信号分配给单个细胞并确定细胞类型特征需要自动化解决方案。目前大多数峰值排序方法至少需要部分人工干预(Marre等 人,2012;Prentice等 人,2011),这在高密度阵列场景中难以实施。本文介绍了一套开源、无监督、可扩展的计算工具组,能够对大规模高密度记录进行峰值排序与细胞类型分类。通过将分析流程与实验方案整合,我们既验证了统计方法的可靠性,又实现了对密集网络中特定细胞类型的光遗传学操控。
先前研究已详细阐述了基于波形分析的细胞类型分类方法,但缺乏实验验证。为解决这一问题并提供该分类确实与细胞类型相关的生物学证据,我们采用病毒策略与光遗传学技术相结合的方法选择性激活PV中间神经元,同时对整个网络进行记录。对光遗传学刺激试验的分析表明,被归类为抑制性的细胞在光遗传学刺激期间更易出现放电频率的改变。
我们发现,在代表峰值半高宽和峰峰值的两个维度上进行聚类,能够以较高成功率实现细胞类型分类。通过从PV阳性转染细胞中采样电极,我们将其动作电位与其他细胞类型区分开来。我们的工作证明,细胞外记录能够为构成复杂网络的不同神经元群体提供深入见解。该研究流程促进了病毒策略、光遗传学和电生理记录的整合应用,从而通过操纵复杂网络来更好地理解每种细胞类型的功能贡献。
尽管SpikeMAP是当前唯一能够在高密度多电极阵列上实现推定兴奋性和抑制性神经元鉴定的已知方法(表 1),但其在脉冲排序流程(图 1)中包含的若干环节与现有方法存在重叠,因为这些步骤是执行E/I鉴定前的必要流程。为方便用户将SpikeMAP与现有工具包集成,我们提供模块化协议套件,使得E/I鉴定可与初步脉冲排序步骤分离执行。通过这种方式,用户可先使用Kilosort或其他软件包完成脉冲排序,再将数据导入SpikeMAP进行E/I鉴定。
皮层抑制性神经元具有异质性,快速放电的PV中间神经元作为最大群体展现出窄峰电位的标志性特征,这一特性使其成为验证我们分类方案的理想候选者。然而未来研究可纳入不同类型的抑制性中间神经元,如生长抑素(SOM)和血管活性肠肽(VIP)神经元,以完善抑制性细胞类型的分类体系。另一研究方向可将SpikeMAP应用于人工生成的峰电位数据(Buccino和Einevoll,2021;Laquitaine等 人,2024)。但需要指出的是,仅依靠细胞外波形来区分所有抑制性细胞类型可能难以实现。尽管存在这一局限,本文所呈现的渐进式进展对于深化认知和完善分析方法至关重要。鉴于体外峰电位排序特征可能不同于体内特性(Henze等 人,2000;Nowak等 人,2003;González-Burgos等 人,2005),未来研究需验证这些分类/波形特征能否推广到行为个体,例如通过使用四极电极、光极或植入式电极阵列进行验证。
总之,我们的研究提出了一种完全集成的方法:通过开源、无监督且可扩展的峰电位分类分析流程进行细胞类型分类,并结合利用稳定阶跃函数视蛋白的实验方案,实现了对大规模微电极阵列记录数据的分类验证。这些工具共同构建了一个研究框架,可用于探索基因定义的神经元群体在大规模神经动力学中的活动机制。
¶ 材料与方法
¶ 动物
PV-CRE小鼠(PvalbCre基因编号008069)购自美国杰克逊实验室(位于缅因州巴港)。所有小鼠品系均为C57BL/6x129S4背景的纯合子,饲养在12:12 小时明暗循环环境中,自由获取食物和水。所有实验操作均按照渥太华大学动物护理与兽医服务部批准的规程和指南(协议编号3471)进行。
¶ 立体定位注射
对于立体定位注射,P21小鼠注射0.05 mg/kg丁丙诺啡并通过吸入异氟烷麻醉。使用10 μL汉密尔顿注射器配合33号针头进行注射,每个坐标点注射1 μL。以前囟点为基准的立体定位坐标如下:mPFC [前后位(AP), +2.4—>+2.6;内外侧(ML), ±0.42;背腹侧(DV), –1.5—>–1.8]。DV坐标从软脑膜表面开始计算。为表达ChR2,每个位点(双侧)注射400 nL pAAV-Ef1a-DIO hChR2(C128S/D156A)-EYFP病毒液,离体电生理记录前留出2-3 周恢复期。
¶ 切片
从35至42日龄PV-CRE小鼠中制备前额叶皮层切片,这些小鼠在立体定位注射后2-3 周进行取材。制备切片时,通过吸入异氟烷(百特公司)麻醉小鼠并采用断头法处死。取出脑后,将脑组织浸入冰浴的氯化胆碱基础切割液(成分为:119 mM 氯化胆碱、2.5 mM KCl、1 mM CaCl、4.3 mM MgSO·7HO、1mM NaHPO、1.3 mM L-抗坏血酸钠、26.2 mM NaHCO及11 mM葡萄糖)中进行冠状脑切片。该切割液持续通入95% O2与5% CO2混合气。切片在含标准人工脑脊液(aCSF,成分为:119 mM NaCl、2.5mM CaCl、1.3 mM MgSO·7HO、1 mM NaHPO、26.2 mM NaHCO及11 mM葡萄糖)的恢复槽中于37 °C下复苏,持续通入95% O与5% CO混合气。切片在恢复槽中复苏1 小时,记录前平衡至∼25 °C。实验过程中组织持续灌注新鲜aCSF。本文所示记录时长为24 分钟,具体序列如下:基线(2 分钟)、光刺激开启(2 分钟)、光刺激关闭(2 分钟)、CNQX洗入(5 分钟)、CNQX +下光刺激 开启(2 分钟)、CNQX +下光刺激 关闭(2 分钟)、PTX洗入(5 分钟)、CNQX与PTX +下光刺激 开启(2 分钟)、CNQX与PTX +下光刺激 关闭(2 分钟)。每个切片加载前均记录30 秒MEA芯片基线(无切片)作为对照。切片在多电极阵列上进行成像,并留存用于病毒表达部位的免疫组化验证。
¶ 全细胞电生理学
采用正置显微镜对神经元进行全细胞记录可视化:BX61WU正置显微镜(60 倍,1.0 NA物镜;奥林巴斯,梅尔维尔,纽约)。通过EYFP表达视觉识别PV神经元。全细胞记录使用硼硅酸盐玻璃膜片电极(3-6MΩ;World Precision Instruments)进行,电极通过Narishige PC-10拉针仪制备。电信号通过Axon Multiclamp 700B放大器记录,以2 kHz滤波,并通过Axon Digidata 1440 A数字化仪以10 kHz采样率数字化。所有实验在室温下于林格氏液中完成,该溶液成分包含(单位mM):119 NaCl、2.5 CaCl2、1.3 MgSO4·7H2O、1 NaH2PO4、26.2 NaHCO3及11葡萄糖,溶液经95% O2和5% CO2饱和(pH 7.3,渗透压295–310mOsm/L)。电极内充灌细胞内液,其组成为(单位mM):115 K-葡萄糖酸盐、20 KCl、10 HEPES、4ATP-Mg、0.5 GTP、10 Na-磷酸肌酸(pH = 7.2–7.3;渗透压270–290 mOsm/L)。
¶ 多电极阵列记录
细胞外电压记录采用瑞士3Brain公司(https://www.3brain.com/)的高密度多电极阵列系统完成。该系统的技术细节可参阅Imfeld等 学者2008年的论文及Berdondini等 学者2009年的研究。通过BioCAM X平台搭载HD-MEA Arena芯片(3Brain GmbH,瑞士),对小鼠前额叶皮层进行了体外高密度记录。该系统采用4096个方形微电极(间距=42 μμm)以64×64布局排列(记录区域2.67mm×2.67 mm),当全阵列记录时采样频率fs为18 kHz。每通道活动记录分辨率为12位,数据通过3Brain提供的BrainWaveX软件进行可视化与采集。
¶ 脉冲排序
SpikeMAP的Python和Matlab代码库已作为开源软件在figshare发布,同时也可从GitHub获取(存档副本见Giraud, 2020)。
¶ 过滤
多电极阵列记录的细胞外电压通常反映靠近特定电极的多个细胞的动态(Rey等人,2015),这引发了如何分离单个单元的问题。单个通道的未滤波电压特征表现为急剧偏转(图2Bi),这是自发性活跃皮层回路体外细胞外活动的典型特征(Hemberger等人,2019)。作为分离单个单元的必要第一步,我们采用二阶巴特沃斯带通滤波器(300–5000 Hz;图2Bii)对细胞外电压进行滤波,以去除缓慢变化的场电位和高频噪声(Bullmann等人,2019)。记录伪迹(电压偏转≥500 μV)被移除并设置为平均电压值。
¶ 尖峰检测
假定的尖峰时间采用电压阈值方法(Lewicki, 1998)进行检测,通过识别滤波信号均值以下x个标准差(SD)的负信号峰值。此处x针对每个记录通道单独计算,通过绘制各通道滤波电压直方图并拟合高斯曲线,以确定出现非正常假定尖峰相关活动的SD值。应用了绝对不规则期,剔除 <2毫秒内的连续事件。随后提取每个尖峰时刻前后(–5至 +5毫秒)的未滤波电压(图 2Bii),得到各通道一系列高频(18 kHz)波形。本文所示数据中,三个记录均采用x =4.5 SD阈值。
¶ 单个单元分离
为分离单细胞活动,我们对各通道进行了主成分分析,结果显示存在对应于单个细胞的独特聚类簇(图2E)。随后采用k均值聚类算法(k=2;Xu与Wunsch,2005)从rrSVD的主成分中提取聚类簇。对标记后的主成分应用线性判别分析(LDA;Hill等 学者,2011)以评估该方法的分离效果。LDA在假定类别协方差相等的前提下,可估算两个聚类簇之间的线性分类错误率。通过十折交叉验证进行评估,其中0.5的错误率对应随机分类水平(图 2G)。对于错误率低于随机水平的聚类簇,我们通过统一标记所有波形进行合并,每个聚类簇被视为一个推定神经元。
¶ 质心三角剖分
为了精确估计每个记录电极之间的胞体位置,我们采用了质心分析方法(Muthmann等 人,2015;Hilgen等 人,2017)。针对先前识定的每个假定胞体,通过提取相邻电极上的波形构建了3×3矩阵,其元素为aij。该矩阵位置(2,2)对应胞体电极平均波形中负峰的振幅值,周围位置则填充与胞体电极直接相邻的所有电极的负峰振幅值。质心的行坐标由以下公式给出:
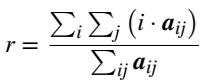
而该列是:
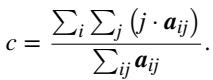
除了质心三角定位之外,SpikeMAP 还包括执行单极三角定位和基于网格的卷积的协议,提供基于波形幅度估计假定的胞体位置的额外选项。
¶ 样条插值
每个单细胞的平均波形使用分段三次样条插值(Matlab R2015b)以更高的速率重采样,通过使用90 kHz的查询点在一组已知点的边界内构建新点。
¶ E/I 分类
我们采用细胞类型特异性病毒策略与光遗传学技术相结合的方法来区分细胞类型。将稳定阶跃功能视蛋白(pAAV-Ef1a-DIO hChR2(C128S/D156A)-EYFP)双侧注射至PV-CRE小鼠(B6.Cg-Pvalb<tm1.1(cre)Aibs>/J)的内侧前额叶皮层(mPFC;Berndt等 人,2009)。该方法可通过100毫秒465 纳米蓝光脉冲选择性长效激活小白蛋白(PV)中间神经元,并通过10 秒545-580 纳米绿黄光脉冲实现失活。随后我们计算了每个神经元的光刺激诱发放电频率变化的z值,以基线均值的标准差为单位表示(图 5D)。我们筛选出放电频率显著增加的细胞(z值≥ >3),通过此方法共分类(n==8189,3张切片,来自3 只小鼠)。总体而言,7759个细胞(94.75%)被可靠地分类为兴奋性神经元,430个细胞(5.25%)被可靠鉴定为抑制性神经元。该数值接近皮层中PV中间神经元的预期占比(4-6%;Markram等 人,2004)。
¶ LED光刺激
LED光刺激通过PlexBright LED模块及控制器(465 纳米;Plexon)进行。光线经由200 μ微米贴片光纤传输,光纤尖端裸露,暴露玻璃部分长度为∼1 厘米。光灭活操作采用黄色激活宽带545-580 纳米滤光片(BP545-580;奥林巴斯)实现。
¶ 免疫组织化学
用于免疫组织化学的脑切片取自35至42日龄小鼠,所有溶液均以1×PBS作为溶剂。小鼠通过吸入异氟烷麻醉,随后经心脏灌注5 mL pH=7.4的PBS,再灌注10 mL 4%多聚甲醛/0.1 M PBS(pH 7.4)持续10 分钟。在4% PFA中进行2 小时固定后,于2天内依次进行15%和30%蔗糖梯度冷冻保护。脑组织在−30 °C异戊烷中冷冻,包埋于冷冻基质,经冷冻切片机(CM 3050 S; Leica)切成40 μμm切片。自由漂浮切片于0.5% PBS-Triton-X-100(TX-100)中封闭45 分钟,并在20 °C条件下于封闭液中孵育GFP一抗(GFP-1020;1:1000, AvesLabs)。次日晨,切片经0.5% PBS-Triton-X-100洗涤三次(每次5 分钟),随后在室温下孵育山羊抗鸡AlexaFluor 488标记二抗(Jackson Labs, 703-546-155)3 小时。切片用0.1% Tx-100洗涤三次(每次5 分钟),最后用Vectashield(Vector Labs)封片。所有用于记录的切片均另存于pH=7.4的10%福尔马林PBS溶液中,以备免疫组化验证病毒表达,实验流程同上。
¶ 附加文件
¶ 补充文件 MDAR 清单
¶ 数据可用性
本研究期间生成或分析的所有数据均包含于稿件、支持文件及在线补充数据中。
生成了以下数据集:
| 作者 | 年份 | 数据集标题 | 数据集网址 | 数据库和标识符 |
|---|---|---|---|---|
| 蒂维耶热 JP | 2025 | SpikeMAP:高密度多电极阵列中用于皮层兴奋性和抑制性神经元的无监督锋电位分类流程 | https://doi.org/10.6084/m9.figshare.28458749.v1 | figshare, 10.6084/m9.figshare.28458749.v1 |
| 蒂维耶热 JP | 2025 | 来自四只小鼠的高密度多电极记录数据集 | https://doi.org/10.6084/m9.figshare.29416472 | figshare, 10.6084/m9.figshare.29416472 |
¶ 参考文献
Bakkum DJ, Obien MEJ, Radivojevic M, Jäckel D, Frey U, Takahashi H, Hierlemann A. 2019. The axon initial segment is the dominant contributor to the neuron’s extracellular electrical potential landscape. Advanced Biosystems 3:e1800308. DOI: https://doi.org/10.1002/adbi.201800308, PMID: 30882024
Barthó P, Hirase H, Monconduit L, Zugaro M, Harris KD, Buzsáki G. 2004. Characterization of neocortical principal cells and interneurons by network interactions and extracellular features. Journal of Neurophysiology 92:600–608. DOI: https://doi.org/10.1152/jn.01170.2003, PMID: 15056678
Becchetti A, Gullo F, Bruno G, Dossi E, Lecchi M, Wanke E. 2012. Exact distinction of excitatory and inhibitory neurons in neural networks: a study with GFP-GAD67 neurons optically and electrophysiologically recognized on multielectrode arrays. Frontiers in Neural Circuits 6:63. DOI: https://doi.org/10.3389/fncir.2012.00063, PMID: 22973197
Berdondini L, Imfeld K, Maccione A, Tedesco M, Neukom S, Koudelka-Hep M, Martinoia S. 2009. Active pixel sensor array for high spatiotemporal resolution electrophysiological recordings from single cell to large scale neuronal networks. Lab on a Chip 9:2644–2651. DOI: https://doi.org/10.1039/b907394a, PMID: 19704979
Berndt A, Yizhar O, Gunaydin LA, Hegemann P, Deisseroth K. 2009. Bi-stable neural state switches. Nature Neuroscience 12:229–234. DOI: https://doi.org/10.1038/nn.2247, PMID: 19079251
Blanche TJ, Swindale NV. 2006. Nyquist interpolation improves neuron yield in multiunit recordings. Journal of Neuroscience Methods 155:81–91. DOI: https://doi.org/10.1016/j.jneumeth.2005.12.031, PMID: 16481043
Buccino AP, Einevoll GT. 2021. MEArec: a fast and customizable testbench simulator for ground-truth extracellular spiking activity. Neuroinformatics 19:185–204. DOI: https://doi.org/10.1007/s12021-020-09467-7, PMID: 32648042
Bugeon S, Duffield J, Dipoppa M, Ritoux A, Prankerd I, Nicoloutsopoulos D, Orme D, Shinn M, Peng H, Forrest H, Viduolyte A, Reddy CB, Isogai Y, Carandini M, Harris KD. 2022. A transcriptomic axis predicts state modulation of cortical interneurons. Nature 607:330–338. DOI: https://doi.org/10.1038/s41586-022-04915-7, PMID: 35794483
Bullmann T, Radivojevic M, Huber ST, Deligkaris K, Hierlemann A, Frey U. 2019. Large-scale mapping of axonal arbors using high-density microelectrode arrays. Frontiers in Cellular Neuroscience 13:404. DOI: https://doi.org/10.3389/fncel.2019.00404, PMID: 31555099
Buzsáki G. 2004. Large-scale recording of neuronal ensembles. Nature Neuroscience 7:446–451. DOI: https://doi.org/10.1038/nn1233, PMID: 15114356
Buzsáki G. 2010. Neural syntax: cell assemblies, synapsembles, and readers. Neuron 68:362–385. DOI: https://doi.org/10.1016/j.neuron.2010.09.023, PMID: 21040841
Calinski T, Harabasz J. 1974. A dendrite method for cluster analysis. Communications in Statistics - Theory and Methods 3:1–27. DOI: https://doi.org/10.1080/03610927408827101
Cardin JA, Carlén M, Meletis K, Knoblich U, Zhang F, Deisseroth K, Tsai LH, Moore CI. 2009. Driving fast-spiking cells induces gamma rhythm and controls sensory responses. Nature 459:663–667. DOI: https://doi.org/10.1038/nature08002, PMID: 19396156
Carlson D, Carin L. 2019. Continuing progress of spike sorting in the era of big data. Current Opinion in Neurobiology 55:90–96. DOI: https://doi.org/10.1016/j.conb.2019.02.007, PMID: 30856552
Chung JE, Magland JF, Barnett AH, Tolosa VM, Tooker AC, Lee KY, Shah KG, Felix SH, Frank LM, Greengard LF. 2017. A fully automated approach to spike sorting (Mountainsort). GitHub. https://github.com/flatironinstitute/mountainsort
Cohen L, Mizrahi A. 2015. Plasticity during motherhood: changes in excitatory and inhibitory layer 2/3 neurons in auditory cortex. The Journal of Neuroscience, 35:1806–1815. DOI: https://doi.org/10.1523/JNEUROSCI.1786-14.2015. PMID: 25632153
Connors BW, Gutnick MJ. 1990. Intrinsic firing patterns of diverse neocortical neurons. Trends in Neurosciences, 13:99–104. DOI: https://doi.org/10.1016/0166-2236(90)90185-d. PMID: 1691879
Constantinidis C, Goldman-Rakic PS. 2002. Correlated discharges among putative pyramidal neurons and interneurons in the primate prefrontal cortex. Journal of Neurophysiology, 88:3487–3497. DOI: https://doi.org/10.1152/jn.00188.2002. PMID: 12466463
Delgado Ruz I, Schultz SR. 2014. Localising and classifying neurons from high density MEA recordings. Journal of Neuroscience Methods, 233:115–128. DOI: https://doi.org/10.1016/j.jneumeth.2014.05.037. PMID: 24954540
Fiscella M, Farrow K, Jones IL, Jäckel D, Müller J, Frey U, Bakkum DJ, Hantz P, Roska B, Hierlemann A. 2012. Recording from defined populations of retinal ganglion cells using a high-density CMOS-integrated microelectrode array with real-time switchable electrode selection. Journal of Neuroscience Methods, 211:103–113. DOI: https://doi.org/10.1016/j.jneumeth.2012.08.017. PMID: 22939921
Giraud E. 2020. BRWExtract. Software Heritage. https://archive.softwareheritage.org/swh:1:dir:b951d5ffe9d07cff91f2ddf631b40a8121ba211d;origin=https://github.com/evgiraud/BRWExtract;visit=swh:1:snp:b6ea2c549be6339a1e08a06af6b384d1bfb91bcd;anchor=swh:1:rev:c8610c979368bbe52b871b0175f814d4030a75ba
González-Burgos G, Krimer LS, Povysheva NV, Barrionuevo G, Lewis DA. 2005. Functional properties of fast spiking interneurons and their synaptic connections with pyramidal cells in primate dorsolateral prefrontal cortex. Journal of Neurophysiology, 93:942–953. DOI: https://doi.org/10.1152/jn.00787.2004. PMID: 15385591
Hafizi H, Nigam S, Barnathan J, Ren N, Stevenson IH, Masmanidis SC, Newman EL, Sporns O, Beggs JM. 2022. Inhibition-dominated rich-club shapes dynamics in cortical microcircuits. bioRxiv. DOI: https://doi.org/10.1101/2021.05.07.443074
Hasenstaub A, Shu Y, Haider B, Kraushaar U, Duque A, McCormick DA. 2005. Inhibitory postsynaptic potentials carry synchronized frequency information in active cortical networks. Neuron, 47:423–435. DOI: https://doi.org/10.1016/j.neuron.2005.06.016. PMID: 16055065
Hemberger M, Shein-Idelson M, Pammer L, Laurent G. 2019. Reliable Sequential activation of neural assemblies by single pyramidal cells in a three-layered cortex. Neuron, 104:353–369. DOI: https://doi.org/10.1016/j.neuron.2019.07.017. PMID: 31439429
Henze DA, Borhegyi Z, Csicsvari J, Mamiya A, Harris KD, Buzsáki G. 2000. Intracellular features predicted by extracellular recordings in the hippocampus in vivo. Journal of Neurophysiology, 84:390–400. DOI: https://doi.org/10.1152/jn.2000.84.1.390. PMID: 10899213
Hilgen G, Sorbaro M, Pirmoradian S, Muthmann JO, Kepiro IE, Ullo S, Ramirez CJ, Puente Encinas A, Maccione A, Berdondini L, Murino V, Sona D, Cella Zanacchi F, Sernagor E, Hennig MH. 2017. Unsupervised spike sorting for large-scale, high-density multielectrode arrays (HS2). GitHub. https://github.com/mhhennig/HS2
Hill DN, Mehta SB, Kleinfeld D. 2011. Quality metrics to accompany spike sorting of extracellular signals. The Journal of Neuroscience, 31:8699–8705. DOI: https://doi.org/10.1523/JNEUROSCI.0971-11.2011. PMID: 21677152
Imfeld K, Neukom S, Maccione A, Bornat Y, Martinoia S, Farine PA, Koudelka-Hep M, Berdondini L. 2008. Large-scale, high-resolution data acquisition system for extracellular recording of electrophysiological activity. IEEE Transactions on Bio-Medical Engineering, 55:2064–2073. DOI: https://doi.org/10.1109/TBME.2008.919139. PMID: 18632369
Insel N, Barnes CA. 2015. Differential activation of fast-spiking and regular-firing neuron populations during movement and reward in the dorsal medial frontal cortex. Cerebral Cortex, 25:2631–2647. DOI: https://doi.org/10.1093/cercor/bhu062. PMID: 24700585
Jia X, Siegle JH, Bennett C, Gale SD, Denman DJ, Koch C, Olsen SR. 2019. High-density extracellular probes reveal dendritic backpropagation and facilitate neuron classification. Journal of Neurophysiology, 121:1831–1847. DOI: https://doi.org/10.1152/jn.00680.2018. PMID: 30840526
Jun J. 2017. JRCLUST. GitHub. https://github.com/JaneliaSciComp/JRCLUST
Jun JJ, Mitelut C, Lai C, Gratiy SL, Anastassiou CA, Harris TD. 2017. Real-time spike sorting platform for high-density extracellular probes with ground-truth validation and drift correction. bioRxiv. DOI: https://doi.org/10.1101/101030
Kawaguchi Y, Kubota Y. 1993. Correlation of physiological subgroupings of nonpyramidal cells with parvalbumin- and calbindinD28k-immunoreactive neurons in layer V of rat frontal cortex. Journal of Neurophysiology, 70:387–396. DOI: https://doi.org/10.1152/jn.1993.70.1.387. PMID: 8395585
Kawaguchi Y, Kubota Y. 1997. GABAergic subtypes and their synaptic connections in rat frontal cortex. Cerebral Cortex, 7:476–486. DOI: https://doi.org/10.1093/cercor/7.6.476
Kohl MM, Paulsen O. 2010. The roles of GABAB receptors in cortical network activity. Advances in Pharmacology, 58:205–229. DOI: https://doi.org/10.1016/S1054-3589(10)58009-8. PMID: 20655484
Lak A, Okun M, Moss MM, Gurnani H, Farrell K, Wells MJ, Reddy CB, Kepecs A, Harris KD, Carandini M. 2020. Dopaminergic and prefrontal basis of learning from sensory confidence and reward value. Neuron, 105:700–711. DOI: https://doi.org/10.1016/j.neuron.2019.11.018. PMID: 31859030
Laquitaine S, Imbeni M, Tharayil J, Isbister JB, Reimann MW. 2024. Spike sorting biases and information loss in a detailed cortical model. bioRxiv. DOI: https://doi.org/10.1101/2024.12.04.626805
Lee J, Carlson D, Shokri H, Yao W, Goetz G, Hagen E, Batty E, Chichilnisky E, Einevoll G, Paninski L. 2017a. YASS: Yet Another Spike Sorter. bioRxiv. DOI: https://doi.org/10.1101/151928
Lee JH, Carlson D, Shokri H, Yao W, Goetz G, Hagen E, Batty E, Chichilnisky EJ, Einevoll G, Paninski L. 2017b. YASS: yet another spike sorter. GitHub. https://github.com/paninski-lab/yass
Lemon RN, Baker SN, Kraskov A. 2021. Classification of cortical neurons by spike shape and the identification of pyramidal neurons. Cerebral Cortex, 31:5131–5138. DOI: https://doi.org/10.1093/cercor/bhab147. PMID: 34117760
Lewicki MS. 1998. A review of methods for spike sorting: the detection and classification of neural action potentials. Network, 9:R53–R78. DOI: https://doi.org/10.1088/0954-898x/9/4/001. PMID: 10221571
Lindén H, Tetzlaff T, Potjans TC, Pettersen KH, Grün S, Diesmann M, Einevoll GT. 2011. Modeling the spatial reach of the LFP. Neuron, 72:859–872. DOI: https://doi.org/10.1016/j.neuron.2011.11.006. PMID: 22153380
Liu YH, Smith S, Mihalas S, Shea-Brown E, Sümbül U. 2021. Cell-type-specific neuromodulation guides synaptic credit assignment in a spiking neural network. PNAS, 118:e2111821118. DOI: https://doi.org/10.1073/pnas.2111821118. PMID: 34916291
Madisen L, Mao T, Koch H, Zhuo J, Berenyi A, Fujisawa S, Hsu YWA, Garcia AJ, Gu X, Zanella S, Kidney J, Gu H, Mao Y, Hooks BM, Boyden ES, Buzsáki G, Ramirez JM, Jones AR, Svoboda K, Han X, et al. 2012. A toolbox of Cre-dependent optogenetic transgenic mice for light-induced activation and silencing. Nature Neuroscience, 15:793–802. DOI: https://doi.org/10.1038/nn.3078. PMID: 22446880
Magland J, Jun JJ, Lovero E, Morley AJ, Hurwitz CL, Buccino AP, Garcia S, Barnett AH. 2020. SpikeForest, reproducible web-facing ground-truth validation of automated neural spike sorters. eLife, 9:e55167. DOI: https://doi.org/10.7554/eLife.55167. PMID: 32427564
Markram H, Toledo-Rodriguez M, Wang Y, Gupta A, Silberberg G, Wu C. 2004. Interneurons of the neocortical inhibitory system. Nature Reviews. Neuroscience, 5:793–807. DOI: https://doi.org/10.1038/nrn1519. PMID: 15378039
Marre O, Amodei D, Deshmukh N, Sadeghi K, Soo F, Holy TE, Berry MJ. 2012. Mapping a complete neural population in the retina. The Journal of Neuroscience, 32:14859–14873. DOI: https://doi.org/10.1523/JNEUROSCI.0723-12.2012. PMID: 23100409
McCormick DA, Connors BW, Lighthall JW, Prince DA. 1985. Comparative electrophysiology of pyramidal and sparsely spiny stellate neurons of the neocortex. Journal of Neurophysiology, 54:782–806. DOI: https://doi.org/10.1152/jn.1985.54.4.782. PMID: 2999347
Mechler F, Victor JD, Ohiorhenuan I, Schmid AM, Hu Q. 2011. Three-dimensional localization of neurons in cortical tetrode recordings. Journal of Neurophysiology, 106:828–848. DOI: https://doi.org/10.1152/jn.00515.2010. PMID: 21613581
Mitchell JF, Sundberg KA, Reynolds JH. 2007. Differential attention-dependent response modulation across cell classes in macaque visual area V4. Neuron, 55:131–141. DOI: https://doi.org/10.1016/j.neuron.2007.06.018. PMID: 17610822
Moore AK, Wehr M. 2013. Parvalbumin-expressing inhibitory interneurons in auditory cortex are well-tuned for frequency. The Journal of Neuroscience, 33:13713–13723. DOI: https://doi.org/10.1523/JNEUROSCI.0663-13.2013. PMID: 23966693
Muthmann JO, Amin H, Sernagor E, Maccione A, Panas D, Berdondini L, Bhalla US, Hennig MH. 2015. Spike detection for large neural populations using high density multielectrode arrays. Frontiers in Neuroinformatics, 9:28. DOI: https://doi.org/10.3389/fninf.2015.00028. PMID: 26733859
Nowak LG, Azouz R, Sanchez-Vives MV, Gray CM, McCormick DA. 2003. Electrophysiological classes of cat primary visual cortical neurons in vivo as revealed by quantitative analyses. Journal of Neurophysiology, 89:1541–1566. DOI: https://doi.org/10.1152/jn.00580.2002. PMID: 12626627
Obien MEJ, Deligkaris K, Bullmann T, Bakkum DJ, Frey U. 2014. Revealing neuronal function through microelectrode array recordings. Frontiers in Neuroscience, 8:423. DOI: https://doi.org/10.3389/fnins.2014.00423. PMID: 25610364
Pachitariu M, Sridhar S, Stringer C. 2023. Solving the spike sorting problem with kilosort. GitHub. https://github.com/cortex-lab/KiloSort. DOI: https://doi.org/10.1101/2023.01.07.523036
Peters AJ, Fabre JMJ, Steinmetz NA, Harris KD, Carandini M. 2021. Striatal activity topographically reflects cortical activity. Nature, 591:420–425. DOI: https://doi.org/10.1038/s41586-020-03166-8. PMID: 33473213
Pettersen KH, Einevoll GT. 2008. Amplitude variability and extracellular low-pass filtering of neuronal spikes. Biophysical Journal, 94:784–802. DOI: https://doi.org/10.1529/biophysj.107.111179. PMID: 17921225
Peyrache A, Dehghani N, Eskandar EN, Madsen JR, Anderson WS, Donoghue JA, Hochberg LR, Halgren E, Cash SS, Destexhe A. 2012. Spatiotemporal dynamics of neocortical excitation and inhibition during human sleep. PNAS, 109:1731–1736. DOI: https://doi.org/10.1073/pnas.1109895109. PMID: 22307639
Peyrache A, Destexhe A. 2019. Electrophysiological monitoring of inhibition in mammalian species, from rodents to humans. Neurobiology of Disease, 130:104500. DOI: https://doi.org/10.1016/j.nbd.2019.104500. PMID: 31195126
Prentice JS, Homann J, Simmons KD, Tkačik G, Balasubramanian V, Nelson PC. 2011. Fast, scalable, Bayesian spike identification for multi-electrode arrays. PLOS ONE, 6:e19884. DOI: https://doi.org/10.1371/journal.pone.0019884. PMID: 21799725
Ramsay JO, Silverman BW. 1997. Functional Data Analysis. Springer.
Ren N, Ito S, Hafizi H, Beggs JM, Stevenson IH. 2020. Model-based detection of putative synaptic connections from spike recordings with latency and type constraints. Journal of Neurophysiology, 124:1588–1604. DOI: https://doi.org/10.1152/jn.00066.2020. PMID: 32937091
Rey HG, Pedreira C, Quian Quiroga R. 2015. Past, present and future of spike sorting techniques. Brain Research Bulletin, 119:106–117. DOI: https://doi.org/10.1016/j.brainresbull.2015.04.007. PMID: 25931392
Riquelme JL, Hemberger M, Laurent G, Gjorgjieva J. 2023. Single spikes drive sequential propagation and routing of activity in a cortical network. eLife, 12:e79928. DOI: https://doi.org/10.7554/eLife.79928. PMID: 36780217
Rossant C, Kadir SN, Goodman DFM, Schulman J, Hunter MLD, Saleem AB, Grosmark A, Belluscio M, Denfield GH, Ecker AS, Tolias AS, Solomon S, Buzsáki G, Carandini M, Harris KD. 2016. Spike sorting for large, dense electrode arrays. Nature Neuroscience, 19:634–641. DOI: https://doi.org/10.1038/nn.4268. PMID: 26974951
Sakata S, Harris KD. 2009. Laminar structure of spontaneous and sensory-evoked population activity in auditory cortex. Neuron, 64:404–418. DOI: https://doi.org/10.1016/j.neuron.2009.09.020. PMID: 19914188
Shein-Idelson M, Pammer L, Hemberger M, Laurent G. 2017. Large-scale mapping of cortical synaptic projections with extracellular electrode arrays. Nature Methods, 14:882–890. DOI: https://doi.org/10.1038/nmeth.4393. PMID: 28805794
Sirota A, Montgomery S, Fujisawa S, Isomura Y, Zugaro M, Buzsáki G. 2008. Entrainment of neocortical neurons and gamma oscillations by the hippocampal theta rhythm. Neuron, 60:683–697. DOI: https://doi.org/10.1016/j.neuron.2008.09.014. PMID: 19038224
Stark E, Eichler R, Roux L, Fujisawa S, Rotstein HG, Buzsáki G. 2013. Inhibition-induced theta resonance in cortical circuits. Neuron, 80:1263–1276. DOI: https://doi.org/10.1016/j.neuron.2013.09.033. PMID: 24314731
Stringer C, Pachitariu M, Steinmetz NA, Okun M, Bartho P, Harris KD, Sahani M, Lesica NA. 2016. Inhibitory control of correlated intrinsic variability in cortical networks. eLife, 5:e19695. DOI: https://doi.org/10.7554/eLife.19695. PMID: 27926356
Sylwestrak EL, Jo YJ, Vesuna S, Wang X, Holcomb B, Tien RH, Kim DK, Fenno L, Ramakrishnan C, Allen WE, Chen R, Shenoy KV, Sussillo D, Deisseroth K. 2022. Cell-type-specific population dynamics of diverse reward computations. Cell, 185:3568–3587. DOI: https://doi.org/10.1016/j.cell.2022.08.019. PMID: 36113428
Tamura H, Kaneko H, Kawasaki K, Fujita I. 2004. Presumed inhibitory neurons in the macaque inferior temporal cortex: visual response properties and functional interactions with adjacent neurons. Journal of Neurophysiology, 91:2782–2796. DOI: https://doi.org/10.1152/jn.01267.2003. PMID: 14711977
Thivierge JP, Giraud E, Lynn M, Théberge Charbonneau A. 2022. Key role of neuronal diversity in structured reservoir computing. Chaos, 32:113130. DOI: https://doi.org/10.1063/5.0111131. PMID: 36456321
Vendramin L, Campello R, Hruschka ER. 2009. On the comparison of relative clustering validity criteria. Proceedings of the 2009 SIAM International Conference on Data Mining, 733–744. DOI: https://doi.org/10.1137/1.9781611972795.63
Wilson FA, O’Scalaidhe SP, Goldman-Rakic PS. 1994. Functional synergism between putative gamma-aminobutyrate-containing neurons and pyramidal neurons in prefrontal cortex. PNAS, 91:4009–4013. DOI: https://doi.org/10.1073/pnas.91.9.4009
Xu R, Wunsch D. 2005. Survey of clustering algorithms. IEEE Transactions on Neural Networks, 16:645–678. DOI: https://doi.org/10.1109/TNN.2005.845141. PMID: 15940994
Yael D, Bar-Gad I. 2017. Filter based phase distortions in extracellular spikes. PLOS ONE, 12:e0174790. DOI: https://doi.org/10.1371/journal.pone.0174790. PMID: 28358895
Yger P, Spampinato GL, Esposito E, Lefebvre B, Deny S, Gardella C, Stimberg M, Jetter F, Zeck G, Picaud S, Duebel J, Marre O. 2018. A spike sorting toolbox for up to thousands of electrodes validated with ground truth recordings in vitro and in vivo. eLife, 7:e34518. DOI: https://doi.org/10.7554/eLife.34518. PMID: 29557782
Yger P, Stimberg M, Lefebvre B, Gardella C, Marre O, Guzman J, Burman A, Acland B. 2023. SpyKING CIRCUS. GitHub. https://github.com/spyking-circus/spyking-circus/