¶ 脑嵌合体揭示个体对神经毒性诱因的易感性
Nature文章,文章地址:https://doi.0rg/10.1038/s41586-024-07578-8
¶ 简介
个体间的遗传变异影响许多疾病的易感性和进展1,2。然而,研究个体人脑在正常发育和疾病表型方面的差异的努力受到忠实的细胞人类模型的缺乏以及当前系统难以扩展以代表多个人的限制。在此,我们提出人脑嵌合体,这是一种高度可重复的、多供体人脑皮层类器官模型,通过单个类器官中来自多个个体供体的细胞共同发育生成。通过在神经干细胞或神经祖细胞阶段重新聚集来自多个单供体类器官的细胞,我们生成了嵌合体,其中每个供体产生大脑皮层的所有细胞谱系,即使使用具有显著生长偏向的多能干细胞系也是如此。我们使用嵌合体研究对表现出高度临床表型变异的神经毒性触发因素的个体间变异:乙醇和抗癫痫药物丙戊酸。个体供体在对目标细胞类型的影响的显现率以及每种受影响细胞类型内的分子表型方面均存在差异。我们的结果表明,人类遗传背景可能是神经毒素易感性的重要介质,并引入嵌合体作为一种可扩展的系统,用于高通量研究脑发育和疾病过程中个体间的变异。
遗传变异在对疾病诱因的差异易感性中起着至关重要的作用1,2。然而,尽管个体易感性的影响可以在体外系统中检测到 1,3–5,但由于缺乏既忠实于内源性人类生物学又可以在许多人中扩展的实验模型,探索其机制受到阻碍。
在大脑中,开创性的研究已经将多能干细胞(PSC)衍生的二维(2D)神经元培养适应为产生包含许多供体细胞系的群体规模模型6–11。然而,2D培养系统通常产生的细胞类型谱有限,无法反映内源性大脑的多样性。三维(3D)细胞培养模型,如人脑类器官,可以更接近地模拟内源性大脑的细胞复杂性和发育事件。将“盘中村”方法10,11扩展到3D类器官培养的努力受到限制,因为生长速率和分化偏差的差异对类器官系统特别有问题8,在这些系统中,多种细胞类型在长时间内共同发育。事先选择具有匹配生长速率的PSC系可以缓解这个问题11,但限制了实验设计到兼容的细胞系集合。
在此,我们展示了3D多供体嵌合体——一种高度可重复的人类皮质类器官模型,保持了平衡的表示在不同供体的细胞类型中,无需预先选择PSC细胞系。我们使用此模型来测量个体对两种神经毒性应激源的发育暴露的易感性——乙醇(与胎儿酒精综合症相关12)和抗癫痫药物丙戊酸(VPA;与自闭症谱系障碍风险增加相关13,14)。乙醇和VPA都在Chimeroids中的多种细胞类型中引起了变化,重要的是,Chimeroids捕捉到了供体特异性的反应差异。总体而言,我们的研究结果表明,个体间的变异显著影响对神经毒性触发因素的易感性,并且Chimeroids可以用作一个可扩展的平台来测量来自不同人类个体的脑细胞生物反应的变异。
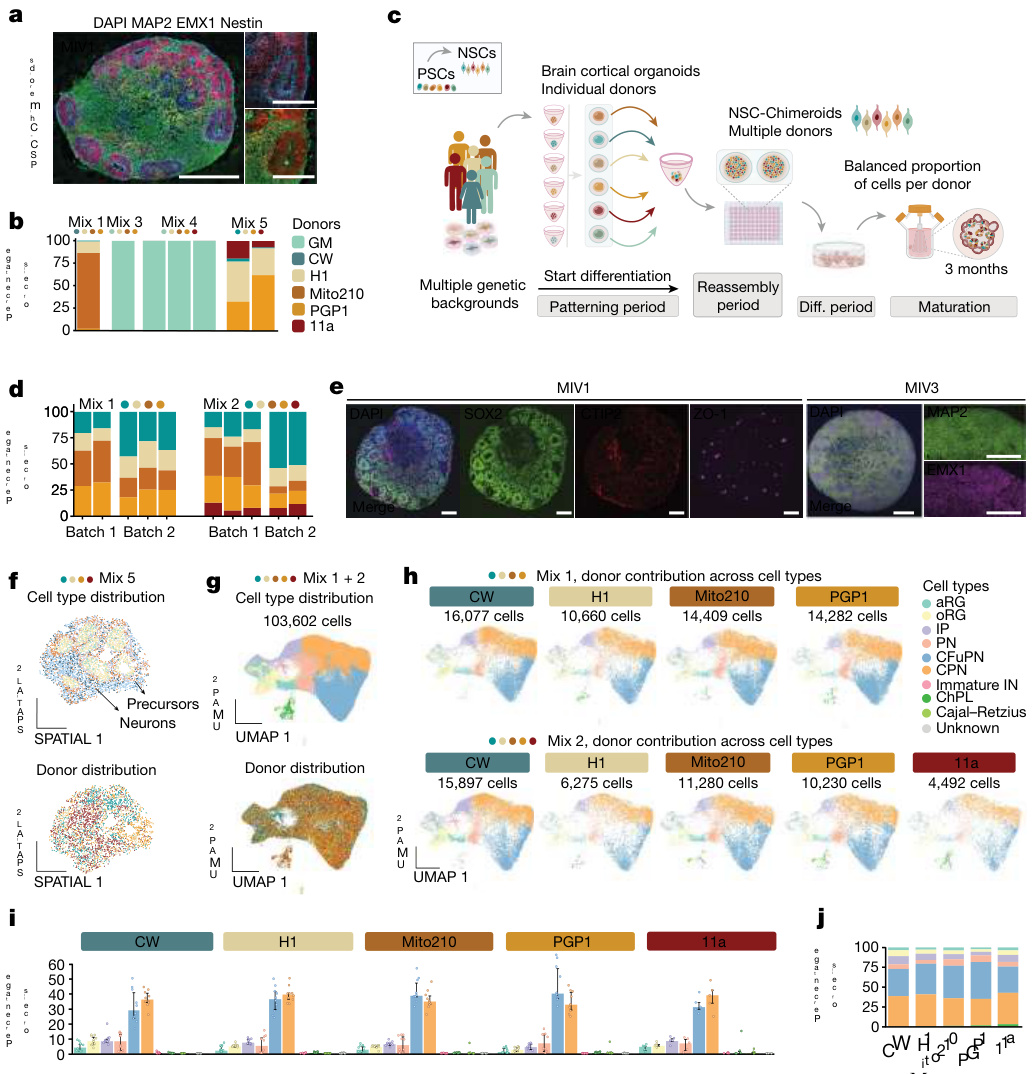
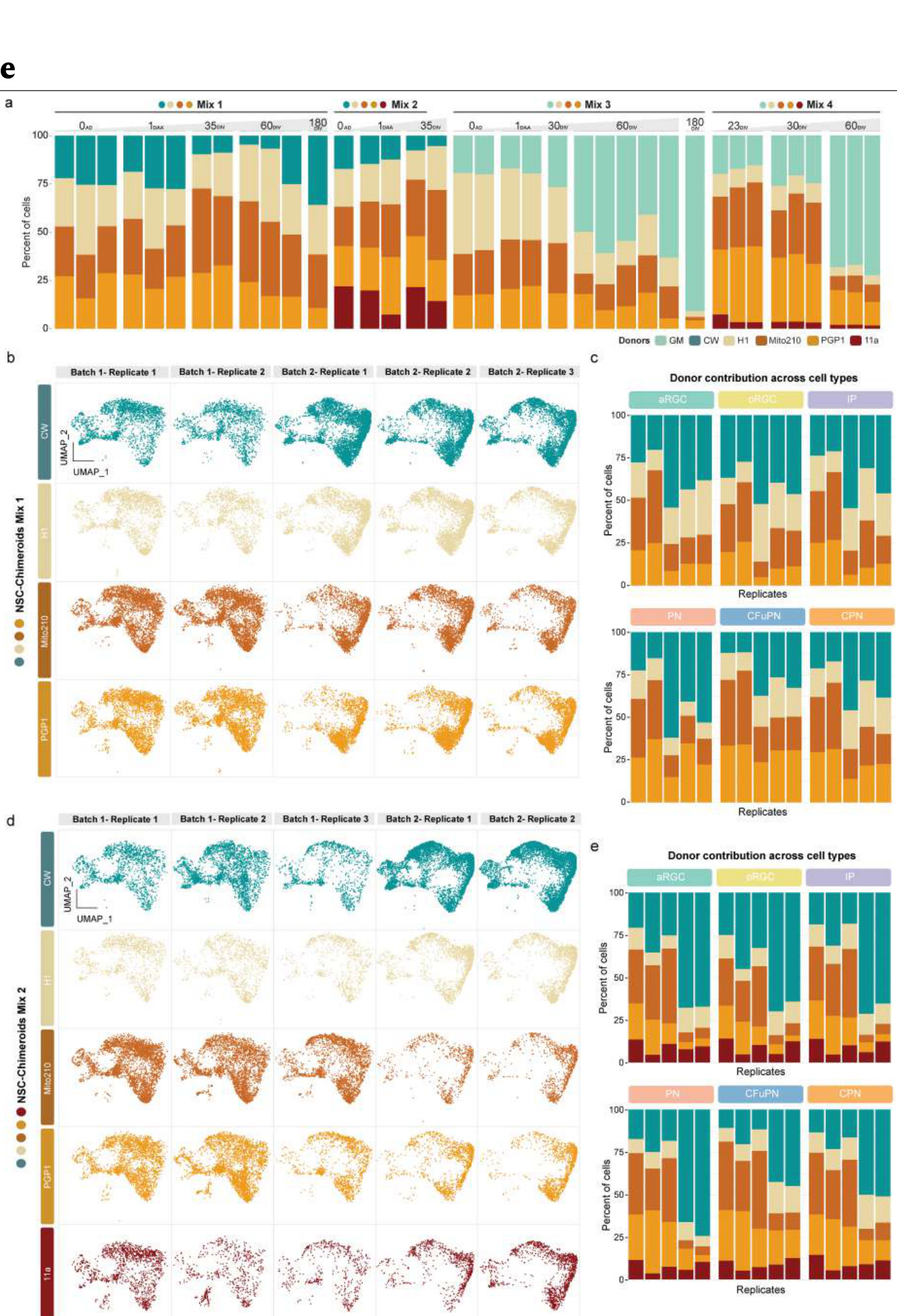
图1 | NSC-Chimeroids 在不同细胞类别中保持供体代表性。a, 在 MIV1 皮层 PSC-Chimeroids 中对 MAP2、EMX1 和 nestin 进行免疫标记。比例尺, (主图)和 2 (插图)。b, MIV3 PSC-Chimeroids 中的供体贡献(解复用的 scRNA-seq),按供体进行颜色编码。b 中使用的供体颜色编码在所有图中均使用。c, NSC-Chimeroid 协议的示意图。Diff. 期,分化 期。部分图标使用 BioRender 创建。d, MIV3 NSC-Chimeroids 中的供体贡献(细胞系丰度),如图所示生成。
在 c. e 中,MIV1 和 MIV3 皮质 NSC-Chimeroids 的免疫标记。比例尺,100 μm(MI V1)和 500 μm(MIV3)。f,MIV2 MD-NSC-Chimeroids(混合 5)的 Slide-seq 分 析,以稳健的细胞类型分解(RCTD)着色。
解离的PSCs在96孔板中混合并聚集形成胚状体;根据我们之前发表的 协议18,胚状体被模式化15-18 天以诱导背侧前脑命运,新生的类器官被 置于CDMII培养基中的动态(搅动)培养中以允许成熟(方法)。我们 使用Census-seq10(方法)量化了每个供体系对每个类器官的贡献。
在 1 月的体外(MIV1),PSC-Chimeroids 显示出个体供体的不平衡 贡献,这在 MIV3 时加剧(图 1b 和扩展数据图 1b)。我们在不同的实 验中测试了四种不同的供体组合(混合 1、混合 3、混合 4 和混合 5; 补充表 1),在不同的时间点,观察到所有情况下的不平衡生长。包含 GM 供体系的 PSC-Chimeroids(混合 3 和混合 4)表现出该系的显著过 度生长,几乎在 MIV2 时完全占据了 Chimeroids(扩展数据图 1b)。在 23 天的体外(DIV23),混合 4 PSC-Chimeroids 中 54% 的细胞来源于 GM(扩展数据图 1b),在 MIV3 时,单细胞 RNA 测序(scRNA-seq) 随后进行监督。
分配的细胞类型(上)和基于Census-seq的供体贡献(下)。g,整合的NSC-Chimer oids的UMAP分析(显示每个数据集的总细胞数),按注释的细胞类型(上)和供体 系划分,并量化供体贡献(下)。h,按供体分割的UMAP,用于两种不同的混合物 (混合物1和混合物2)。ChPL,脉络丛;未成熟IN,未成熟中间神经元; IP,中间 祖细胞;PN, 未指定的投射神经元。 颜色键 适用于所有图形。i,NSC-Chimeroids 中的细胞类型比例,按供体在混合物1和混合物2中进行去混合。数据为跨重复的中 位数和上下四分位数。n = 10(CW,H1,Mito210和PGP1)和n = 5(11a)。j,混 合物1和混合物2中每个供体的细胞类型比例。
通过遗传变异19(方法)进行的供体分配显示,GM 系列在混合 4 PSC-C himeroids 中贡献了几乎所有(>99%)的细胞(图 1b)。在没有 GM 的 混合物中,Mito210 细胞系被过度代表(在 MIV3 的混合 1 PSC-Chimero ids 中占 84% 的细胞;图 1b)。在没有 GM 和 Mito210 的混合 5 PSC-Ch imeroids 中,一个系列(CW)几乎被淘汰(在 MIV3 中的细胞少于 3% ;图 1b 和扩展数据图 1d–f)。因此,在胚状体形成初期混合不同的 P SC 系列会导致个体供体的代表性极不均衡,这与之前在多巴胺能神经 元模型中的观察结果一致8。值得注意的是,当多个供体保持代表性( 混合 5)时,所有保留的供体都能够以较为平衡的方式贡献于所有可识 别的细胞类型,这表明 Chimeroid 方法本身并不改变个体供体的细胞命 运潜力(扩展数据图 1d–i)。
PSCs在命运潜能方面具有高度增殖性和可塑性,这些特性在类器官发 育早期的微小差异可能导致后期时间的不成比例表现。在PSCs经历神经 命运承诺后混合供体系,当它们已经减少的细胞生长和更受限的命运潜力,可能会减少终端供体贡献的变异 性。因此,我们在神经模式化完成后,在神经干细胞(NSC)阶段进行 了供体混合(NSC-Chimeroids)(图 1c)。
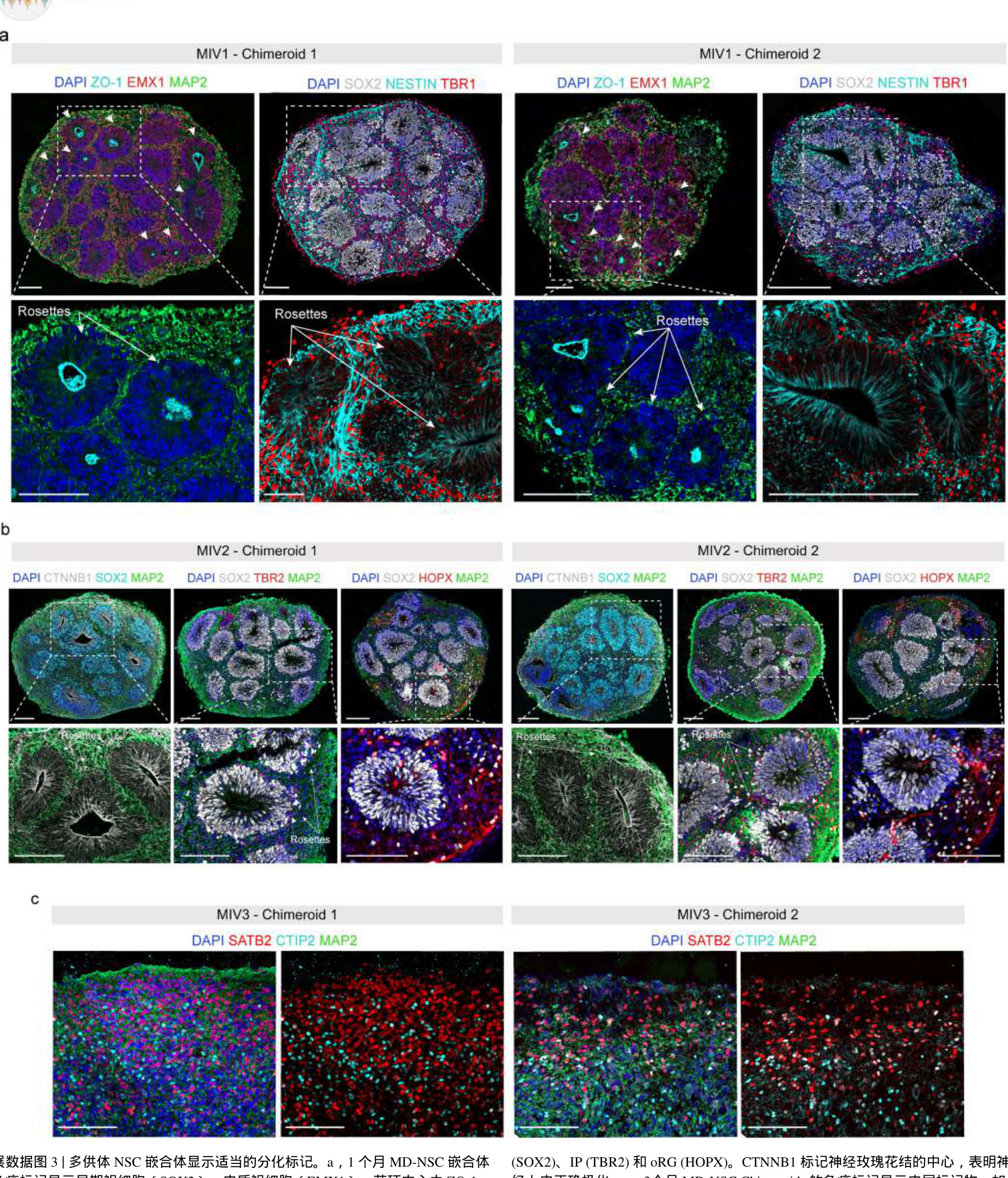
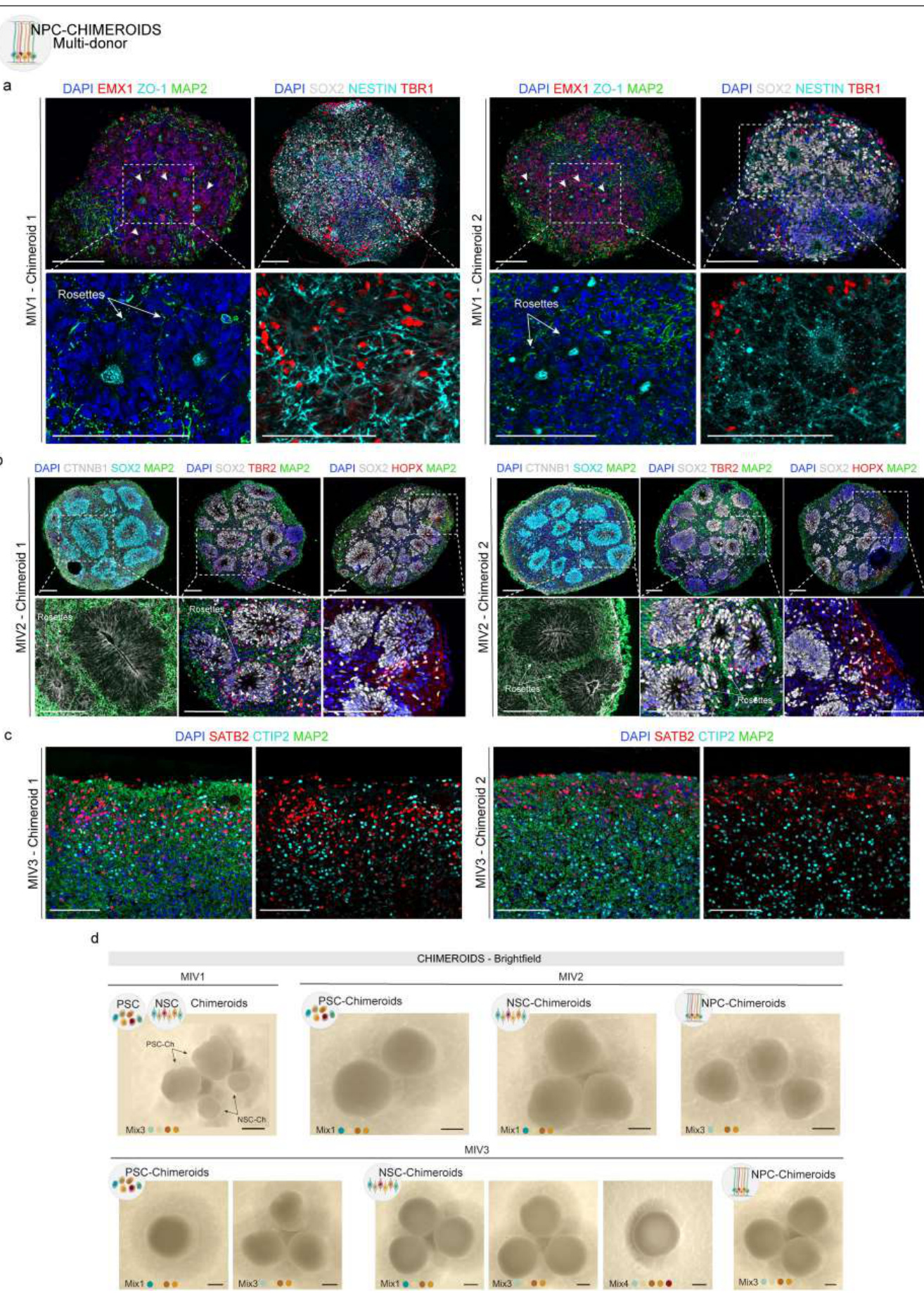
为了创建NSC-Chimeroids,我们首先生成了单供体胚体,并根据我们 之前发布的类器官协议18对其进行模式化,直到DIV15-18。来自4到5个 单独系的初生单供体类器官被解离,以相等的比例混合(聚合第0天) ,在96孔板中以每孔20,000个细胞的密度接种(选择这种细胞密度的原 因在 方法和扩展数据图 2中解释),并允许其再聚合2 天,然后转移到 动态培养中(图 1c和方法)。 在聚集后20 天(DIV35),NSC-Chimeroids已经形成了“花环”—— 由表达端脑标记物FOXG1的细胞、中间祖细胞(IP;TBR2+) 细胞和外 放射状胶质细胞(oRG;HOPX+)细胞组成的前脑室区样(VZ-like)结 构,周围是新生的皮质下投射神经元(CFuPNs; TBR1+CTIP2+)。花 环中心排列着ZO-1,一种存在于放射状胶质(RG)细胞末端的紧密连 接蛋白,以及β-连环蛋白和nestin信号,表明神经上皮极性正确(图 1e 和扩展数据图 3及补充表 2)。
为了创建NSC-Chimeroids,我们首先生成了单供体胚体,并根据我们之前发布的类器官协议18对其进行模式化,直到DIV15-18。来自4到5个单独系的初生单供体类器官被解离,以相等的比例混合(聚合第0天),在96孔板中以每孔20,000个细胞的密度接种(选择这种细胞密度的原因在 方法和扩展数据图 2中解释),并允许其再聚合2 天,然后转移到动态培养中(图 1c和方法)。
在聚集后20 天(DIV35),NSC-Chimeroids已经形成了“ 花环”由表达端脑标记物FOXG1的细胞、中间祖细胞( ) 细胞和外放射状胶质细胞(oRG;HOPX+)细胞组成的前脑室区样(VZ-like)结构,周围是新生的皮质下投射神经元(CFuPNs; TBR1+CTIP2+)。花环中心排列着ZO-1,一种存在于放射状胶质(RG)细胞末端的紧密连接蛋白,以及 -连环蛋白和nestin信号,表明神经上皮极性正确(图 1e和扩展数据图 3及补充表 2)。
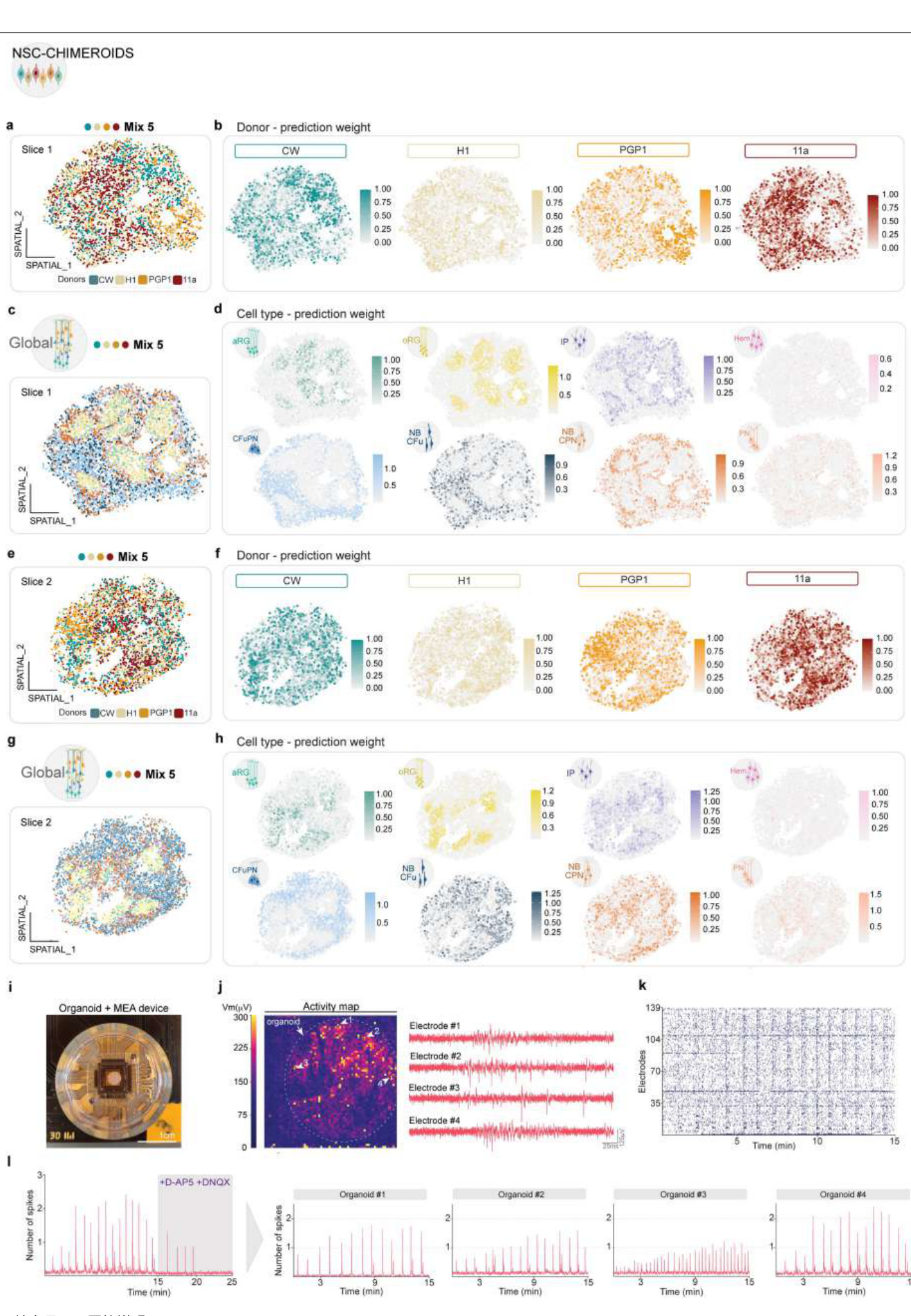
我们使用 Slide-seq 在 DIV55 检查了个体供体的空间组织,其显示的 组织结构与使用免疫组织化学(IHC)观察到的相似(图 1f,方法和扩 展数据图 5a–h)。值得注意的是,来自不同供体的细胞在神经玫瑰体 内大部分是混合在一起的,这表明供体的自我分离并没有驱动类器官结 构。
在不同时间点使用 Census-seq 进行定量分析(n = 37 个重复)(扩展 数据图 4a)和在 MIV3 使用 scRNA-seq(n = 10 个单独的 Chimeroids, 103,602 个细胞;图 1d 和扩展数据图 4b–e)显示,与 PSC-Chimeroids 相比,NSC-Chimeroids 在四种不同混合物中保持了显著更平衡的供体贡 献。值得注意的是,在 PSC-Chimeroids 中由单一供体主导(85% 到 99 %)的混合物 1、3 和 4(图 1b)在 NSC-Chimeroids 中保留了所有供体 的贡献(图 1d)。在 MIV3,NSC-Chimeroids 显示出背侧前脑标志物 的正确表达(图 1e 和扩展数据图 3c)。scRNA-seq 数据显示,在 MIV 3,来自两种不同混合物的 NSC-Chimeroids 生成了我们皮质类器官模型 中预期的完整细胞类型汇编18(图 1g–j)。重要的是,每个供体对 Chi meroids 中的每种细胞类型都有贡献(图 1g–j 和扩展数据图 4b–e) 。
为了评估功能成熟度,我们在 MIV4 时对 mix 2 NSC-Chimeroids 进行 了多电极阵列(MEA)电生理记录,此时该类类器官模型通常具有电 活性1(方法)。我们检测到的网络爆发被 NMDA 和 AMPA 受体拮抗剂 消除,这表明相关活动是由突触谷氨酸传递驱动的,类似于我们原始的 、单一供体的皮质类器官在这个年龄的情况1(扩展数据图 5i–l)。
总之,NSC-Chimeroids 的发育与我们已发表的标准皮层类器官协议 18,20相似,同时保留了代表所有产生的细胞类型中多个供体的能力。
¶ 嵌合体可以控制供体在增殖中的偏差
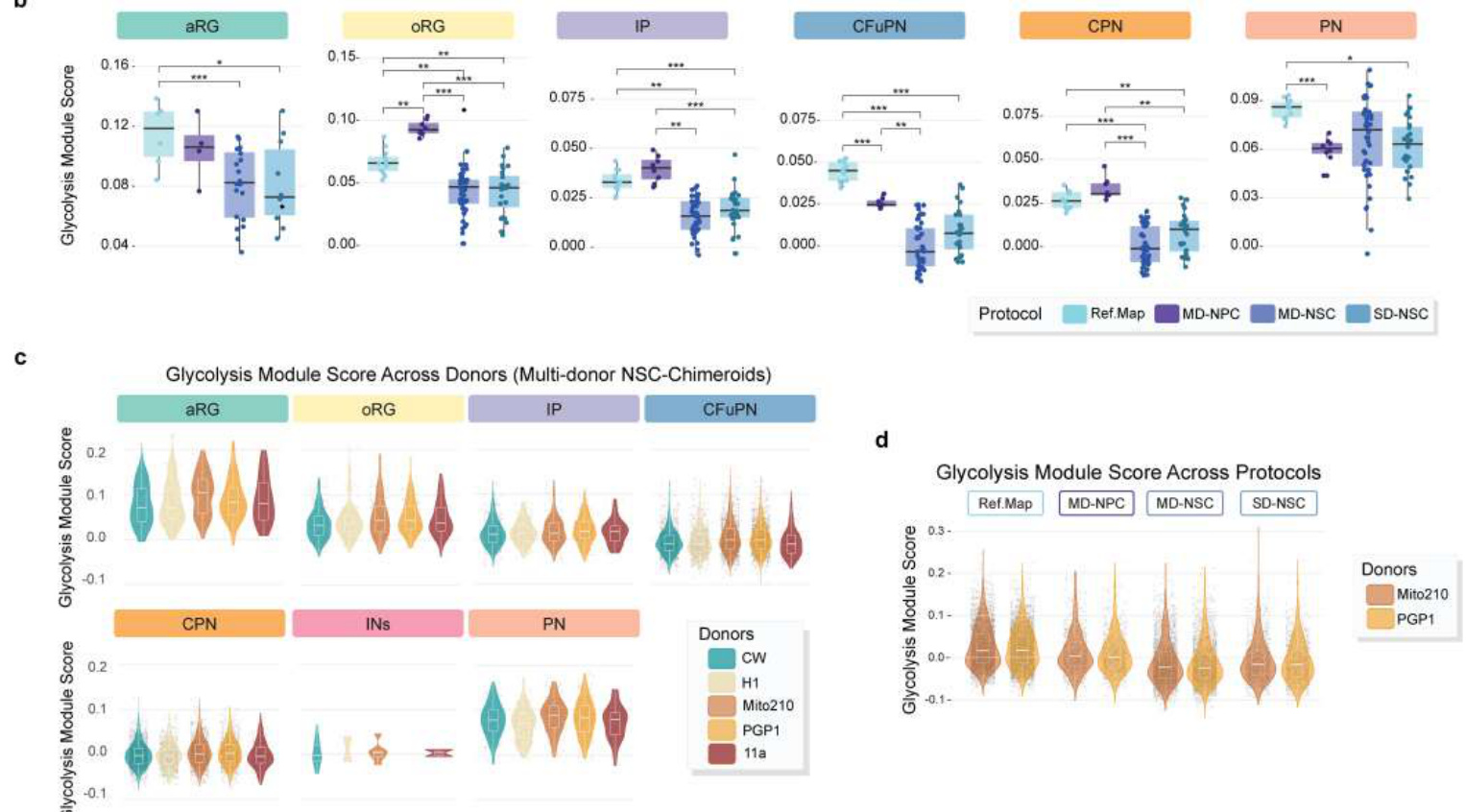
为了研究嵌合体在缓冲供体生长率变异方面的潜力,我们纳入了一条具 有显著增殖优势的PSC细胞系(GM;混合3和混合4;图 1b和补充表 1 )。尽管加入GM确实使NSC-嵌合体中的供体比例发生偏斜(GM贡献 了76–95%的细胞;图 2a–c),但在MIV3阶段,所有供体都得以代表 并能够生成该阶段预期的所有细胞类型20(图 2b–d),这与PSC-嵌合 体形成对比,其中大多数其他细胞系在MIV3阶段几乎或完全被淘汰。 值得注意的是,GM供体系产生了更高比例的神经前体细胞(平均 = 31. 0%,标准差 = 3.28%)与其他供体相比。 (均值 = 13.8%,标准差{s.d. = 6.4%),尤其是oRG细胞(GM:均值 = 16.1%,标准差{s.d. = 0.6%;其他供体:均值 = 6.8%,标准差{s.d. = 2.6 %;图 2e),可能推动了该细胞系的生长优势。伪时间分析同样表明, 与其他供体相比,GM在预测的早期发育阶段的细胞比例增加(图 2e) 。 这一观察表明,在更晚的、增殖性较低的分化和命运承诺阶段混合前 体细胞可能会进一步改善供体生长偏差。因此,我们通过在DIV23-25解 离和混合细胞(NPC-Chimeroids)(图 2f和方法)从神经前体细胞(N PCs)生成嵌合体。
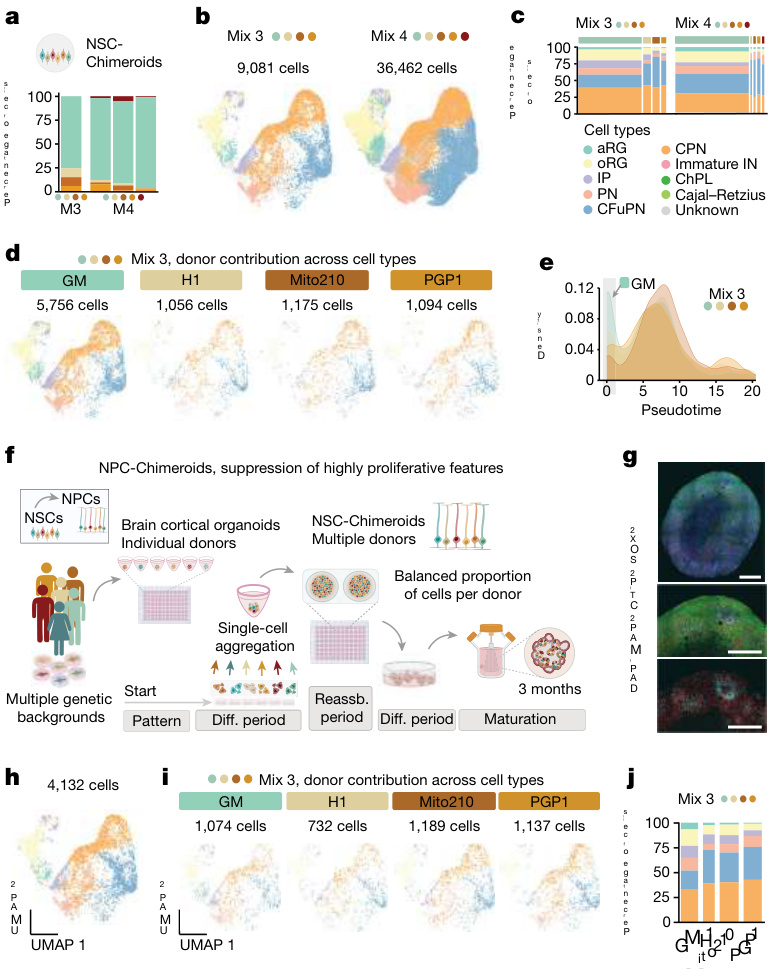
IHC分析MIV1混合3 NPC-Chimeroids确认了神经玫瑰体的形成和预期 的细胞类型标记(扩展数据图 6a)。值得注意的是,在早期时间点(M IV1和MIV2),NPC-Chimeroids整体上比NSC-Chimeroids显得更小,而 PSC-Chimeroids则显得更大。这可能与培养的持续时间有关,因为在MI V1时,PSC-Chimeroids自聚集以来已生长了35 天,而NSC-Chimeroids自 解离-再聚集以来已生长了17-20天,NPC-Chimeroids则生长了10-12天。 然而,在MIV1和MIV2的各个方案中,极性、形态和预期的标记蛋白表 达仍然一致(扩展数据图 1, 3和6)。在MIV3时,大小差异已被平衡,所有的Chimeroid协议在形态或大小上基本上无法区分 (图 2g和扩展数据图 6c,d)。
scRNA-seq在DIV90(n = 2; 4,132个细胞)对NPC-Chimeroids的分析 显示,它们达到了预期的细胞类型组成(图 2h,i),重要的是,即使在 GM系存在的情况下,供体贡献也更加平衡(GM贡献:17–31%(NPC -Chimeroids),76–95%(NSC-Chimeroids)和>99%(PSC-Chimeroids );图 2j)。值得注意的是,GM仍然显示出更高比例的增殖细胞类型 (结合顶端放射状胶质细胞(aRG)、oRG细胞和IPs:GM中为34.4–3 5.5%,而其他供体为11.0–23.8%;二项混合效应模型,P < 10−15;图 2j ),这表明Chimeroid协议保留了供体之间的内在差异。
总体而言,Chimeroid 技术使得在不同发育阶段使用神经祖细胞作为 聚集的起始细胞类型成为可能;在类器官发育的后期进行供体混合可以 克服显著的供体特异性生长优势,而无需事先了解特定细胞系的特性。
¶ 单供体和多供体嵌合体中的一致性细胞多样性
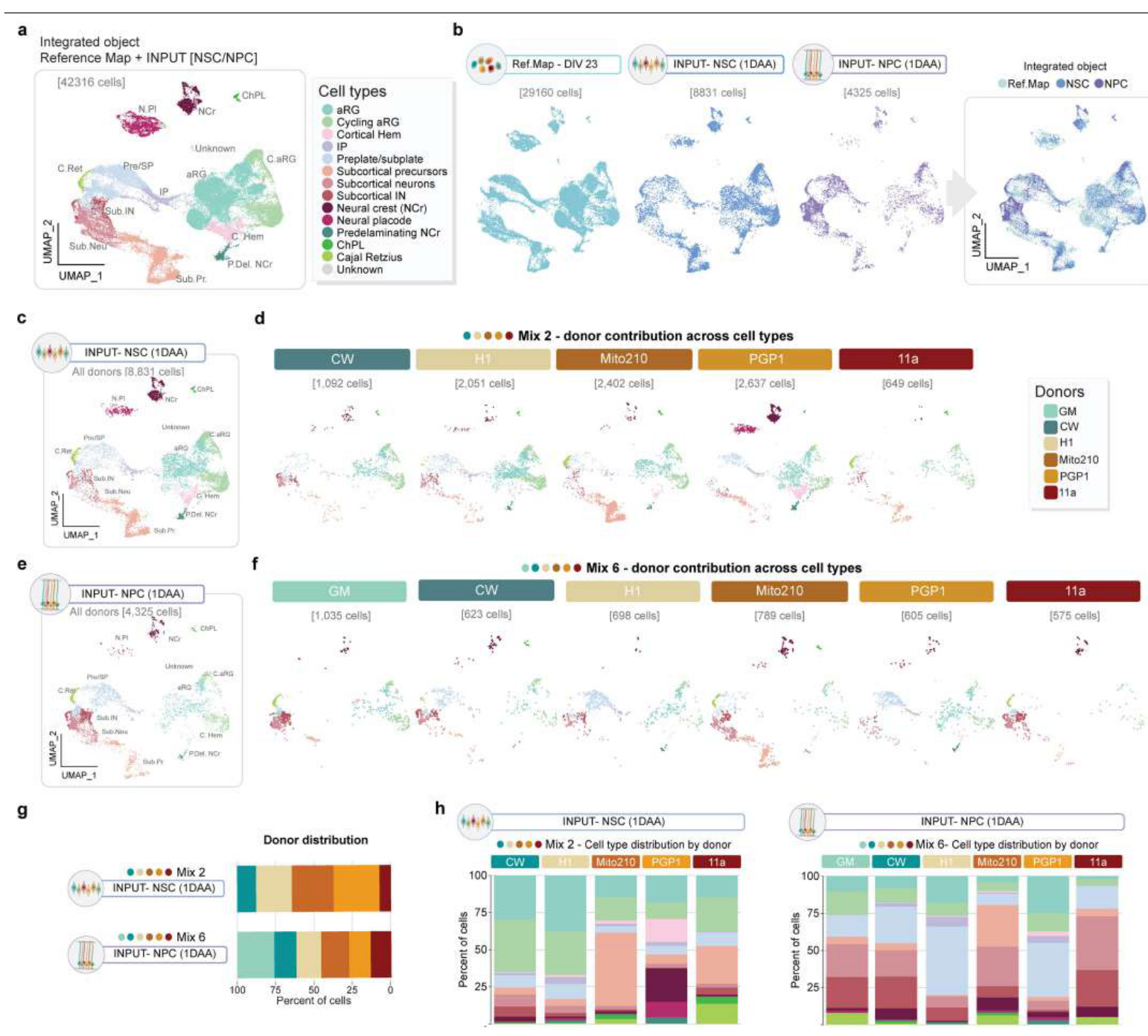
为了了解在不同方案中播种嵌合体的起始细胞类型的身份,我们在解离 -重聚步骤后1 天对每个方案的新生嵌合体进行了分析。我们在聚集后1 天对新重聚的混合2 NSC-嵌合体和混合6 NPC-嵌合体进行了scRNA-seq 分析(n = 10个汇集的嵌合体;8,831个(NSC-嵌合体)和4,325个(NP C-嵌合体)细胞;扩展数据图 7)。两种类型的新生嵌合体都包含前体 细胞(23%和11%的细胞是aRG细胞,分别有2.5%和2.2%是IPs)和早期 生成的神经元(分别为7.0%和23%);这些群体的相对比例在不同方案 中有所不同,这与用作输入的分化阶段相符。两个方案在此阶段都保持 了所有供体的代表性(扩展数据图 7g,h)。
为了研究解离-重聚是否可能改变新生Chimeroids中细胞的分子身份, 我们使用了先前描述系统中建立的类器官发育参考图谱20,该图谱代表 了从第23天到6个月的体外发育时间线(以下简称参考图谱)。与第23 天参考图谱类器官数据的比较显示,重聚后立即出现的细胞类型代表了 这些早期时间点的预期群体(扩展数据图 7),包括在此阶段模型中发 现的早期皮层下命运20(扩展数据图 7h)。数据表明,解离-重聚过程不 会明显 扰乱用于Chimeroid创建的新生类器官中的细胞身份。
为了了解Chimeroid方法是否影响每个供体产生的细胞类型谱,我们 比较了同一供体系在不同模型中产生的细胞组成。作为对照,我们通过 解离和重聚来自单一供体系的新生类器官,生成了单供体(SD)NSC 模拟Chimeroids,并通过在MIV1和MIV3进行IHC分析确认了预期的分 化标记(扩展数据图 8)。
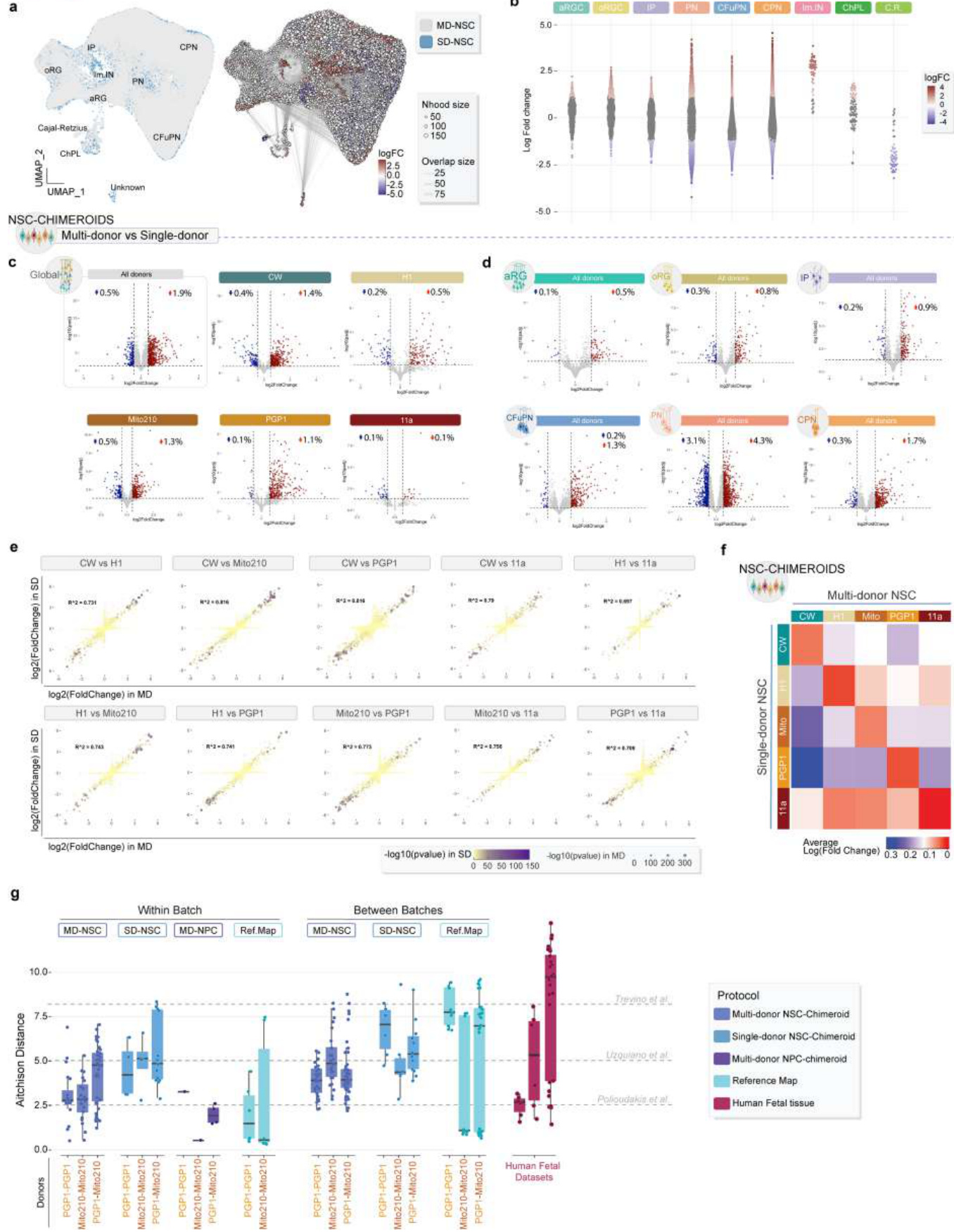
我们通过scRNA-seq在MIV3对来自单供体NSC-Chimeroids(n = 25个 单独的Chimeroids)的54,369个细胞进行了分析,并使用Milo21比较了多 供体NSC-Chimeroids(以下简称MD-NSC-Chimeroids)与单供体NSC-C himeroids(以下简称SD-NSC-Chimeroids)在细胞比例和全局表达谱上 的差异。Milo测试了单细胞数据中差异细胞丰度和表达方差,而不使用 聚类身份。Milo表明,在MD-NSC-Chimeroids和SD-NSC-Chimeroids之 间,表达景观的全局变化最小,离散细胞类型比例的整体变化很小(扩 展数据图 9a,b)。
接下来,我们分析了在 MD-NSC 和 SD-NSC 条件下差异表达的基因 (扩展数据图 9c,d 和补充表 3)。只有一小部分基因(0.6–2.0%)在单 个细胞类型中显著差异表达(扩展数据图 9c,d 和补充表 4)。为了测试 在单供体条件下发现的供体特异性基因表达模式是否也可以在多供体嵌 合体中检测到,我们比较了 SD-NSC 与 MD-NSC 条件下每对细胞系之 间的差异表达基因(DEGs)。供体之间的 DEGs 在不同条件下高度相 关——在 SD-NSC-嵌合体中显著差异表达的大多数基因在 MD-NSC-嵌 合体中也显著(扩展数据图 9e,f)。
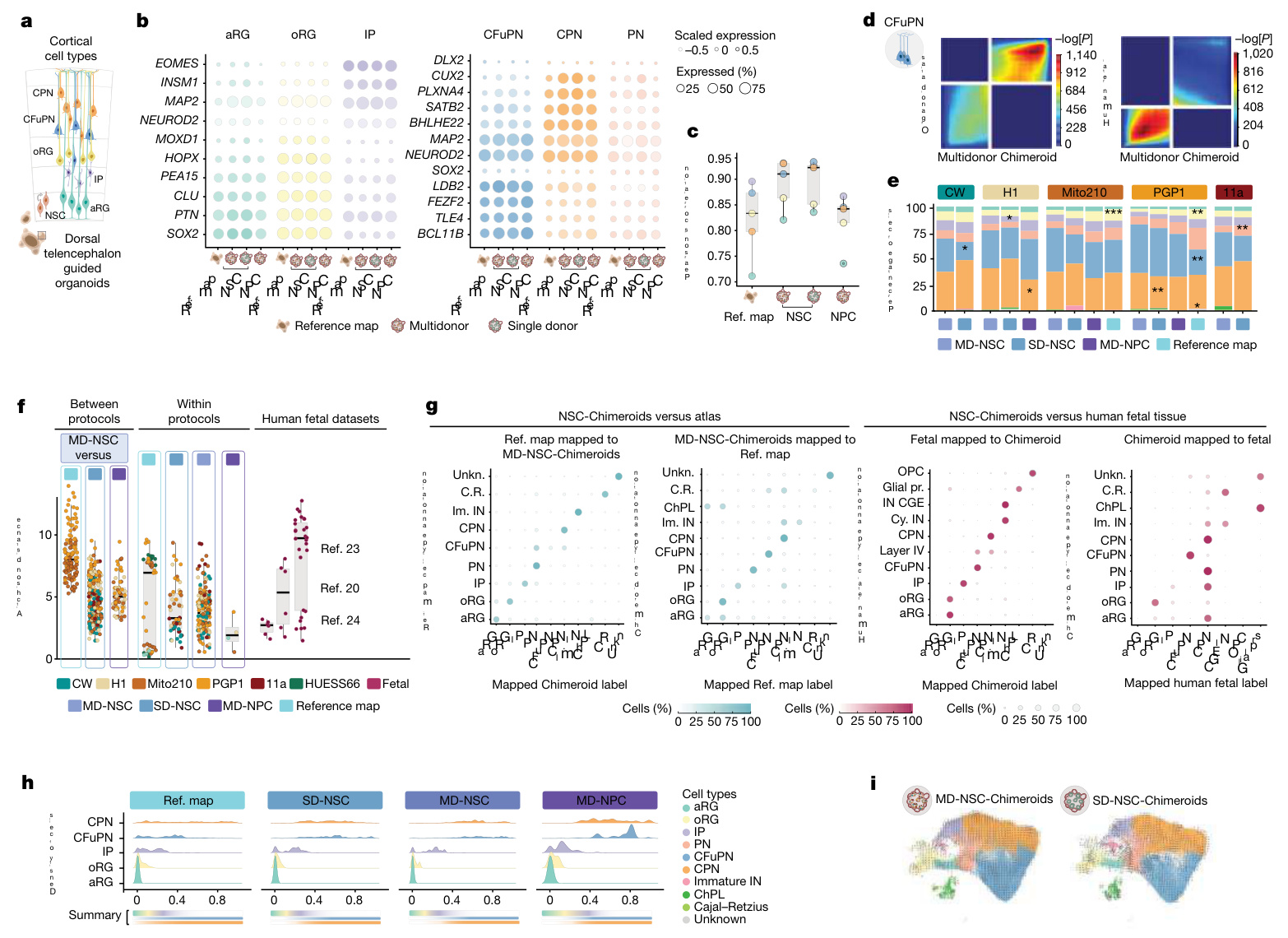
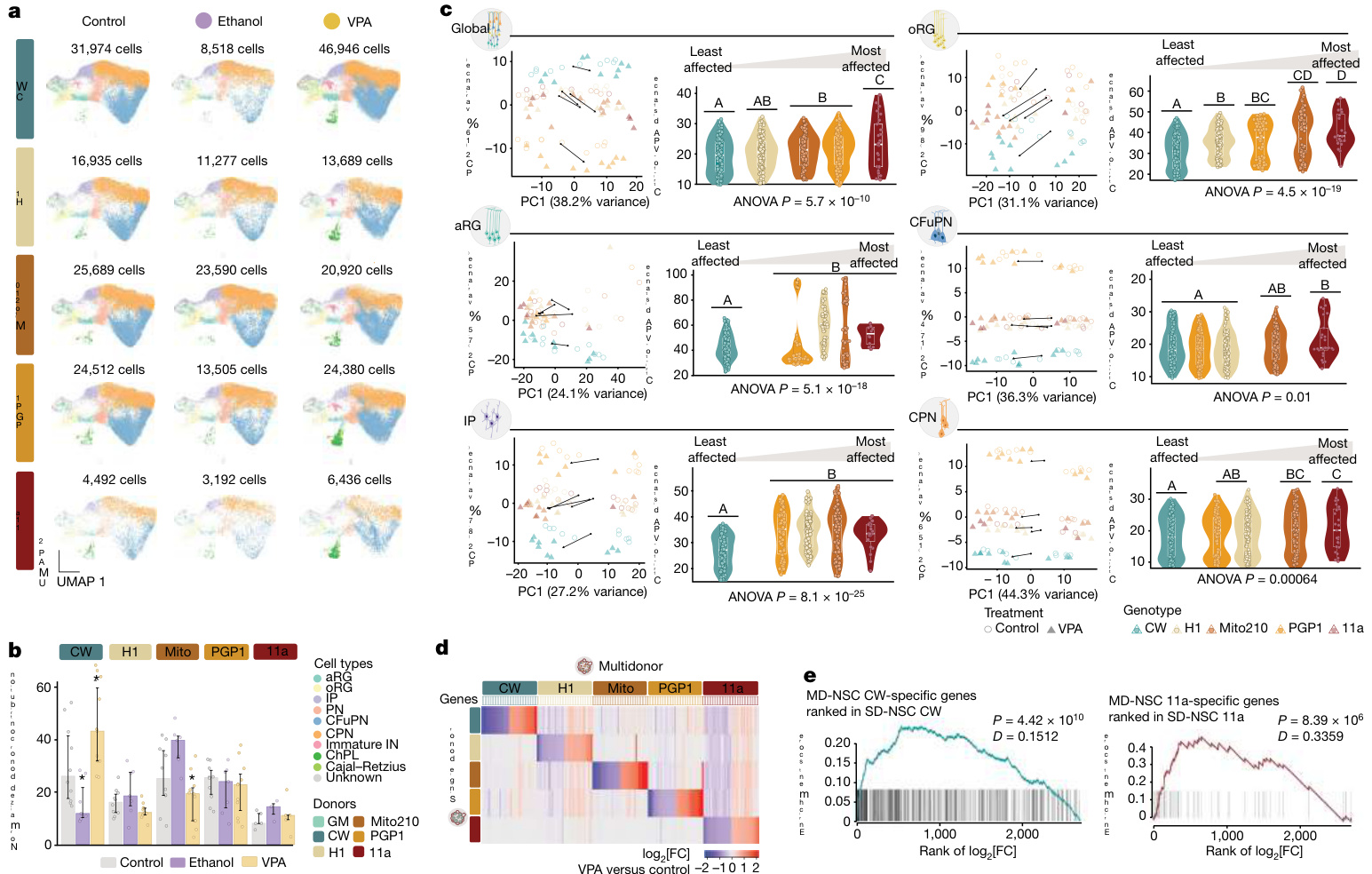
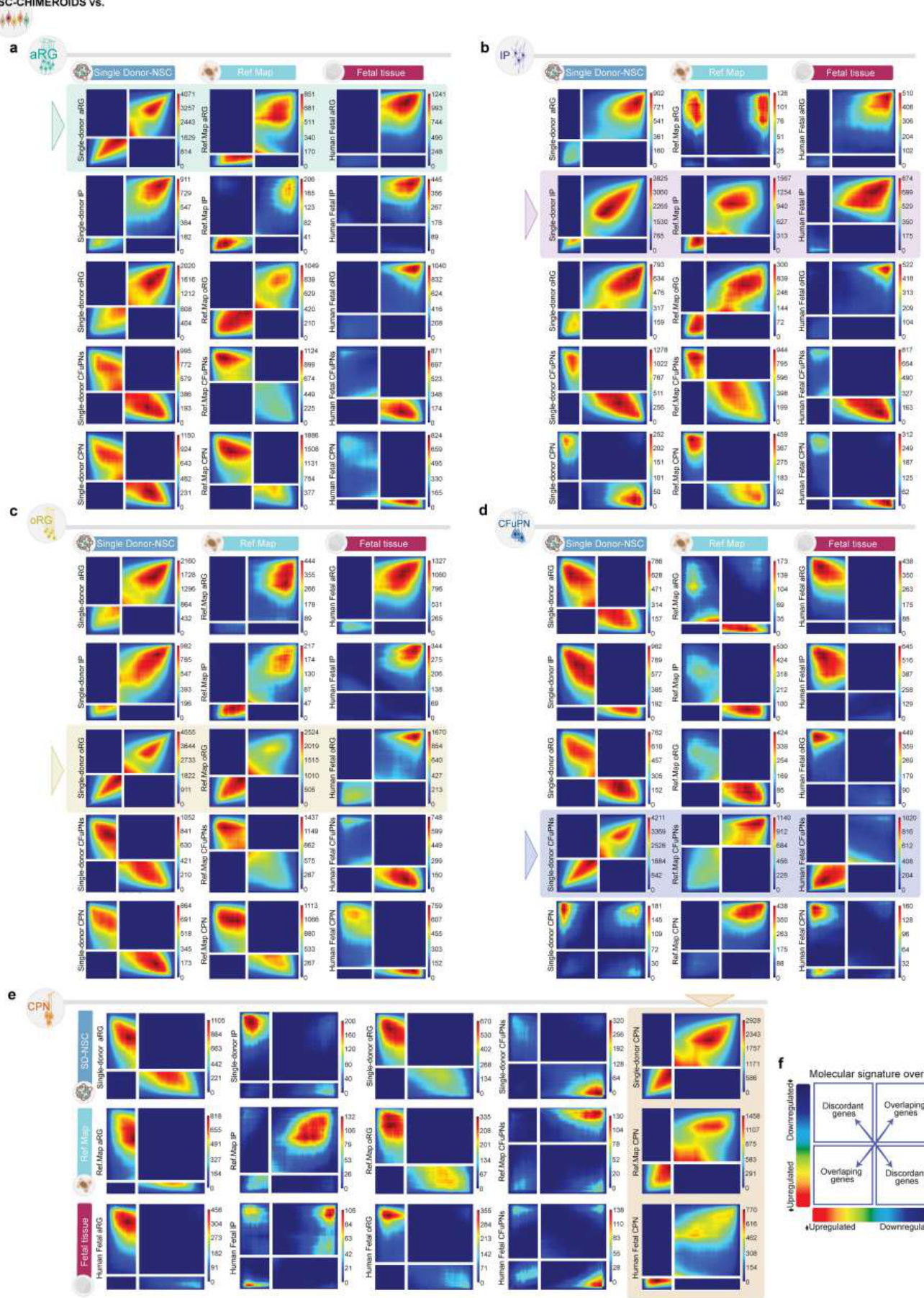
为了测试Chimeroid模型中细胞类型的后续发展是否遵循与我们原始 类器官模型相同的模式,我们验证了所有MIV3 Chimeroid模型在此年龄 段都包含了参考图谱20类器官中存在的相同细胞类型子集(图 3a–c) 。此外,所有Chimeroid模型和参考图谱类器官之间的细胞类型标记基 因高度一致(对应细胞类型之间标准化标记基因表达的Pearson相关系数 范围为0.94–0.99;图 3c和补充表 5)。重要的是,这些标记基因在Chi meroid细胞和我们之前发表的人类胎儿皮层scRNA-seq数据集20(方法) 中的相应内源性细胞类型之间也高度一致(Pearson相关系数范围为0.82 –0.93;图 3c);值得注意的是,MD-NSC-Chimeroids和SD-NSC-Chim eroids与胎儿细胞类型的相关性同样高。对所有表达基因进行的全局转 录相似性评估(秩–秩超几何重叠)22同样显示出MD-NSC-Chimeroids 和SD-NSC-Chimeroids、参考图谱类器官细胞20和内源性胎儿细胞20之间 的高度相似性(图 3d,方法和扩展数据图 10)。
每个供体的细胞类型丰度在各个协议中基本一致(负二项模型;图 3e )。为了更精确地评估细胞类型组成的相对差异,我们计算了协议内和 协议间的Aitchison距离(图 3f和方法),并将其与三个不同人类胎儿皮 层数据集20,23,24中的Aitchison距离进行比较(图 3f)。所有类器官和Chi meroid数据集均在人体胎儿数据集的范围内,表明Chimeroid协议具有高 重现性,可与个体内源性大脑相媲美。重要的是,参考图谱类器官、M D-NSC-Chimeroids和SD-NSC-Chimeroids的协议内距离没有差异(Wilco xon秩和检验,P值从0.42到0.75),这表明在细胞类型组成上的类器官 间变异性在不同协议之间是可比的。
为了比较批次间变异与供体间变异,我们特别检查了在所有 Chimeroi d 协议和参考图集数据集中共享的两条线(方法)——PGP1 和 Mito210 。对于每个供体的批内距离和批间距离以及供体之间的距离,所有比较 的范围都非常相似(扩展数据图 9g)。因此,无论是 Chimeroid 方法还 是在同一系统中结合多个供体都不会增加变异性。
为了确认与内源性人类胎儿皮质细胞类型的转录相似性,我们应用了 参考标签转移25,从NSC-Chimeroids到两个参考数据集中的细胞类型: 内源性人类胎儿皮质20和我们的类器官参考图谱20,反之亦然(方法) 。嵌合体细胞主要被分配到两个数据集中预期的参考细胞类型,再次表 明其表达谱与内源性皮质细胞类型和先前描述的标准类器官18具有高度 相似性(图 3g)。
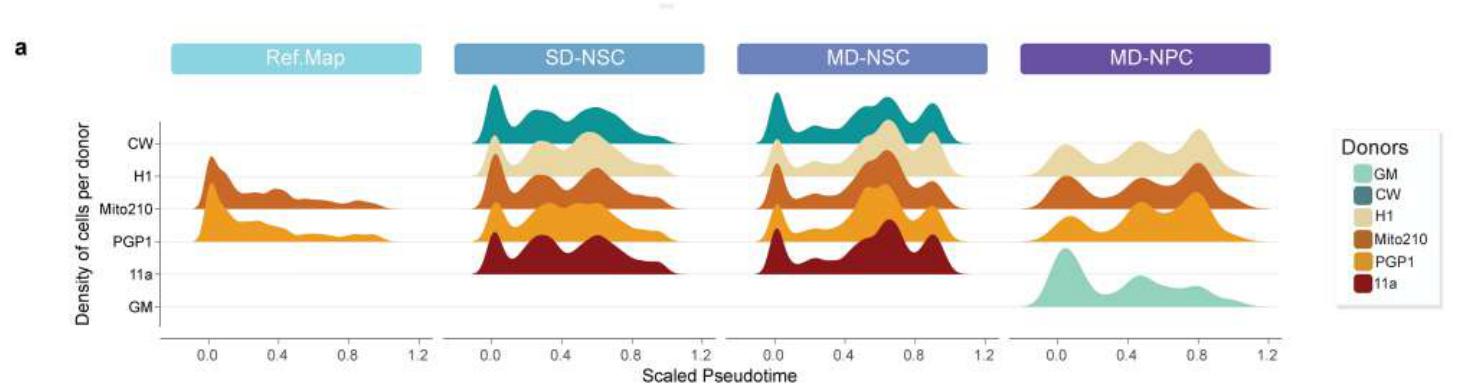
接下来,我们检查了不同协议之间时间轨迹的可能差异。使用 Monoc le 3 (ref. 26)(方法)为单个细胞类型计算的伪时间轨迹显示,参考图谱 类器官和所有 Chimeroid 模型之间的预测发育轨迹相似,主要细胞类型 出现在正确的时间顺序(图 3h)。我们还检查了RNA速度(每个细胞中基因 表达变化的速率)27。基于这种方法,MD-NSC和SD-NSC嵌合体显示出 相似的发展轨迹,并且在每个方案中,个体供体之间高度相似(图 3i和 扩展数据图 11a)。评估糖酵解基因集的表达(扩展数据图 11b–d)显 示,嵌合体方案均未引起细胞应激的显著差异。这些数据表明,多供体 和单供体NSC嵌合体通过相似的分子轨迹发展。
总体而言,这些数据表明,Chimeroid模型在相同供体混合物的重复 实验之间是可重复的,并且是一致的。使用我们已发表的协议1,18,20生产的单供体类器官。MD-NSC-和SD-NSC嵌合体也保留了与参考皮质类器官模型18,20相同程度的细胞复杂性和与 内源性人类胎儿组织的分子相似性。
¶ 乙醇和VPA诱导细胞类型特异性效应
为了应用Chimeroid模型研究个体间对扰动的反应差异,我们测试了两 种可能导致神经发育异常的神经毒性触发因素的影响:乙醇和VPA。人 类胎儿暴露于乙醇可能导致胎儿酒精谱系障碍12,28–36,临床表型32具有高度的个体间变异。用于治疗癫痫和精 神障碍的VPA,在妊娠期间使用时,与后代发展自闭症相关障碍的风险 增加有关13,14。
我们从两种不同的混合物中生成了MD-NSC-Chimeroids,混合物1和 混合物2(补充表 1),并用生理相关浓度的乙醇或VPA处理它们30 天 ,从第45天到第75天(图 4a和方法)。这个时间窗口针对广泛的细胞类 型,包括所有潜在的祖细胞类型以及所有来源于皮层的神经元细胞类型 20。处理后,Chimeroids被培养至DIV90。
我们通过scRNA-seq对来自两个供体混合物的处理过的MIV3 MD-NS C-Chimeroids进行了分析(混合物1,143,591个细胞,n = 15个Chimeroi ds;混合物2,131,636个细胞,n = 13个Chimeroids;图 4b和扩展数据图 12)。来自两个处理组的细胞与未处理的混合物1和混合物2 MD-NSCChimeroids(如上)合并进行聚类和注释(方法)。
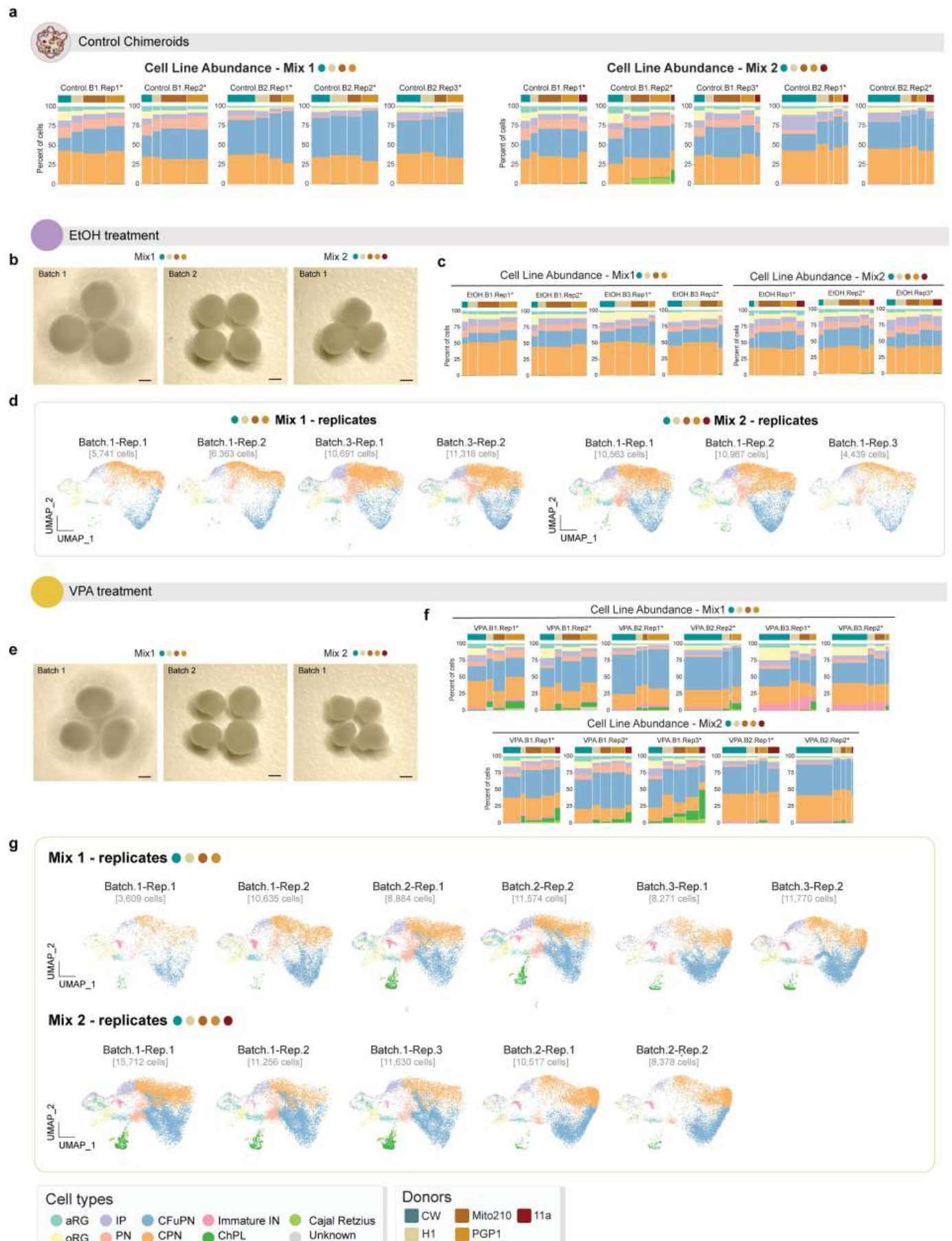
与未经处理的(对照)嵌合体(图 4b)相比,每个处理组在细胞类型 组成上显示出明显且显著的变化(图 4c)。VPA显著增加了未成熟GA BA能中间神经元的比例(平均丰度增加22倍,假发现率(FDR)调整 P = 5.3 × 10−9,负二项混合效应(NBME)模型)和脉络丛细胞(平均 丰度增加6倍,FDR调整P = 1.3 × 10−7),即使在考虑批次、混合和供体 来源的情况下(图 4c,方法和扩展数据图 13a–c),这与先前关于VPA 处理的类器官模型的报告一致37。值得注意的是,我们之前已确定加速 的中间神经元发育是不同自闭症相关障碍相关风险基因中半倍体功能丧 失突变的汇聚表型之一1,这表明该细胞群体可能特别容易受到神经发 育风险因素的影响。此外,胼胝体投射神经元(CPN)和IP的比例均因 乙醇处理而增加(FDR调整P = 0.01(CPN)和0.01(IP),NBME模型 ),而在VPA处理的嵌合体中均减少(FDR调整P = 0.01(CPN)和0.00 35(IP),NBME模型)(图 4c)。
由于单个细胞类型比例的变化是相互依赖的(所有变化的总和必须为 零),我们应用了Milo21,这证实了VPA处理的MD-NSC-Chimeroids中 未成熟中间神经元和脉络丛细胞比例的增加以及IPs的减少,以及乙醇 处理的Chimeroids中CFuPNs比例的减少(图 4e–g)。此外,Milo强调 了表达景观的整体变化,而在VPA处理的Chimeroids中的oRG细胞、CF uPNs和CPNs以及乙醇处理的Chimeroids中的CPNs中,离散细胞类型比 例没有整体变化。在VPA处理的Chimeroids中,17,824个邻域中有6,327 个(35.5%)发生了显著变化,而在乙醇处理的Chimeroids中,13,610个 邻域中有5,589个(41.1%)发生了变化(图 4e–g)。重要的是,我们 观察到VPA处理对SD-NSC-Chimeroids中细胞类型比例的类似影响(图 4d和扩展数据图 13d,e)。
总体而言,这些发现表明,VPA 和乙醇导致了治疗特异性的细胞组 成和细胞内在表达状态的改变,这些改变可以在多供体 Chimeroids 中检 测到,并且与在单供体 Chimeroids 中检测到的表型改变相似,这证实了 使用多供体 Chimeroids 来研究治疗特异性反应的可行性。
¶ 嵌合体识别对神经毒素的个体反应
接下来,我们检查了是否可以检测到Chimeroids对乙醇和VPA处理的供 体特异性反应。虽然每个供体都对所有细胞类型有贡献,无论是否受到 干扰(图 5a),但来自CW供体系的MD-NSC-Chimeroid细胞的比例因 乙醇处理而减少(FDR调整后P = 0.059,NBME)。
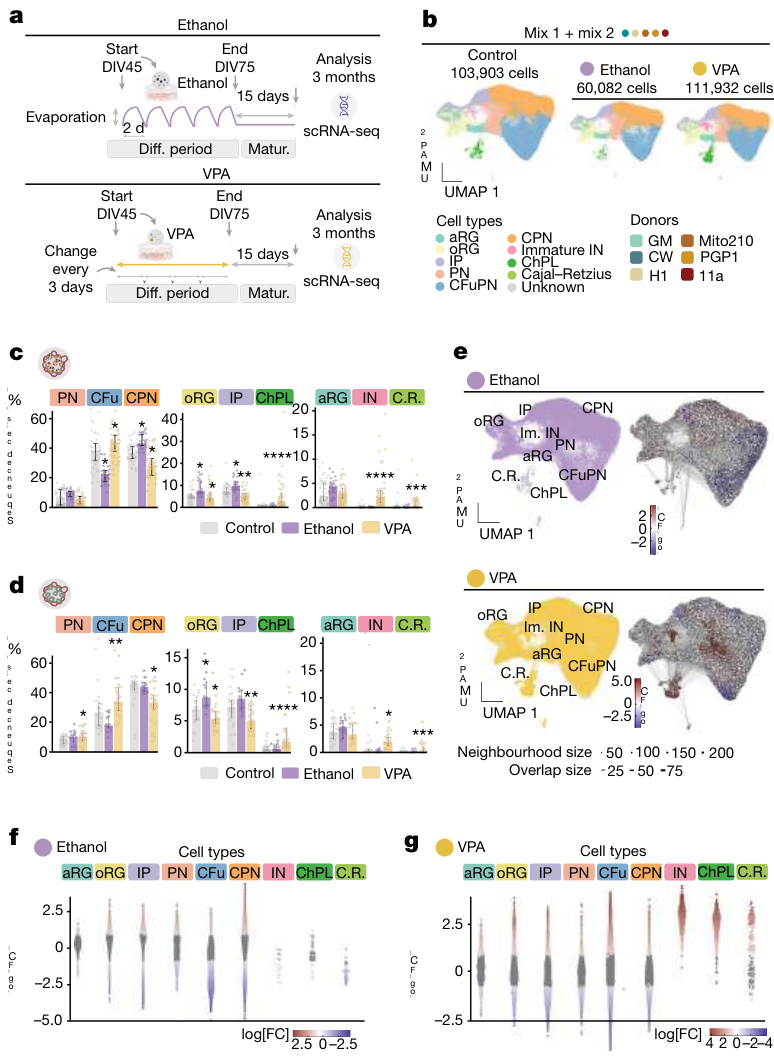
模型)并被 VPA 增加(FDR 调整后的 P = 0.011),表明这两种化合物 具有治疗和供体特异性的效果(图 5b)。 接下来,我们检查了在 VPA 处理的 MD-NSC-Chimeroids 中是否存在 供体特异性的表达响应差异。为了总结每个供体的差异响应幅度,我们 在主成分分析(PCA)后计算了每个供体的伪批量样本之间的欧几里得 距离,既在全局(跨所有细胞类型)上,也在每个主要细胞类型上分别 计算(方法)。 值得注意的是,供体在对VPA的基因表达反应程度上存在差异,无论 是整体上还是在个别细胞类型中(图 5c);对于大多数细胞类型,11a 供体是受影响最显著的,而CW是受影响最小的。重要的是,每个供体 对VPA的反应在单供体和多供体NSC-Chimeroid协议之间通常是一致的 (图 5d和补充表 6和7);我们没有发现任何基因在这两种协议中被VP A显著地以相反方向失调(方法)。
值得注意的是,供体在对VPA的基因表达反应程度上存在差异,无论 是整体上还是在个别细胞类型中(图 5c);对于大多数细胞类型,11a 供体是受影响最显著的,而CW是受影响最小的。重要的是,每个供体 对VPA的反应在单供体和多供体NSC-Chimeroid协议之间通常是一致的 (图 5d和补充表 6和7);我们没有发现任何基因在这两种协议中被VP A显著地以相反方向失调(方法)。
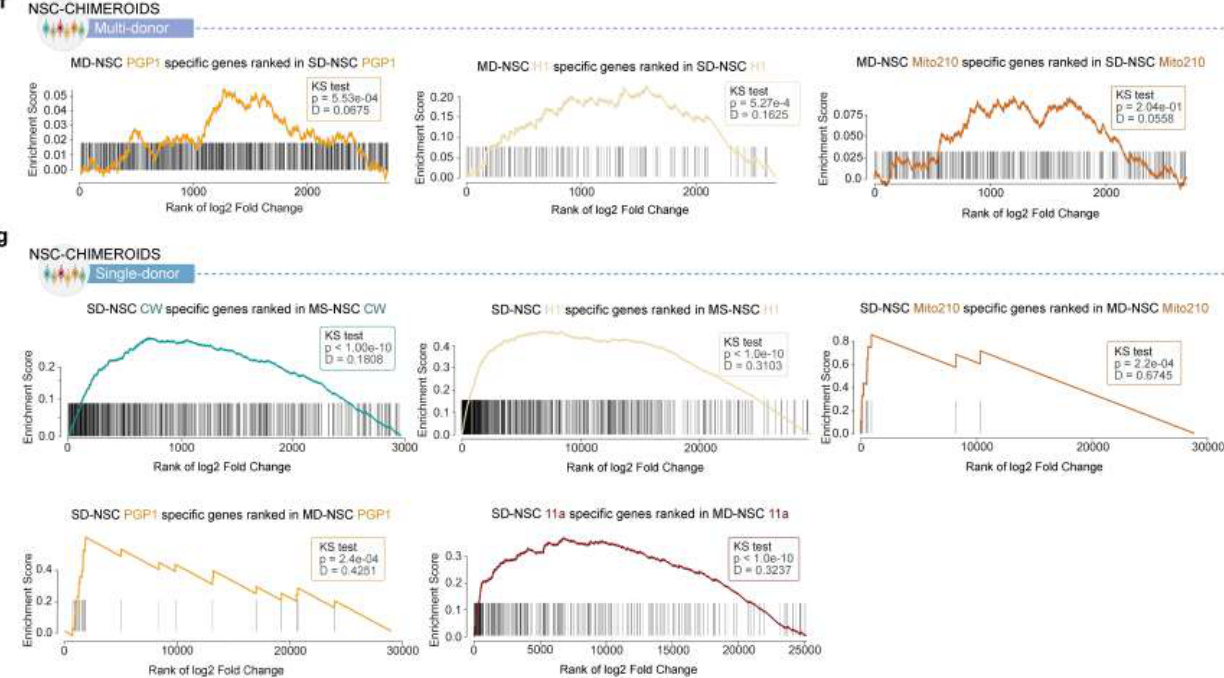
最后,我们确定了在 MD-NSC-Chimeroids 中每个供体特有的 VPA 扰 动基因(图 5d);值得注意的是,受影响较大的供体具有更高比例的 供体特异性。 VPA扰动基因。重要的是,在VPA暴露后,相应的SD-NSC-Chimeroids 中富集了相同的独特基因集(图 5e,扩展数据图 13f,g和补充表 8)。 我们的数据共同表明,多供体Chimeroid系统能够在多个供体中进行 实验扰动的检测,同时仍然允许检测个体反应的差异。数据突显了Chi meroids在高通量研究人类遗传变异对大脑对扰动反应的影响方面的价值 。
¶ 讨论
在人体大脑中模拟个体差异对疾病易感性的贡献一直很困难。并非所有 的人类PSC细胞系在所有体外模型中表现同样出色,这可能与重编程、 表观遗传印记或对培养条件的易感性差异有关。
Chimeroid系统使得在单个类器官中研究来自多个个体的大量不同类 型脑细胞的集体发育和行为成为可能。这种方法将有助于在各种供体特征中进行检测;例如,疾病的遗传风险 ,存在或不存在易感因素,或临床表型。尽管具有这种价值和未来的潜 力,但需要考虑的是,Chimeroid技术劳动强度大,并且确实需要在皮 质类器官系统方面的专业知识才能成功实施;根据我们的经验,成功的 Chimeroid生成依赖于在混合步骤之前验证新生类器官的正确早期模式 。
针对中枢神经系统疾病的治疗通常仅在一部分患者中显示出疗效。未 来的工作旨在扩大供体库,并结合细胞培养系统的自动化程度提高,这 可能使得能够在数百甚至数千个个体中调查群体水平的变异性。使用Ch imeroids进行大规模药物反应检测的能力可能使得能够根据数据将患者 分层到不同的治疗反应组中;随着时间的推移,大型数据集可能会推动 生成模型,旨在在临床研究之前预测药物的疗效。
¶ 在线内容
任何方法、附加参考文献、Nature Portfolio 报告摘要、源数据、扩展数 据、补充信息、致谢、同行评审信息;作者贡献和竞争利益的详细信息 ;以及数据和代码可用性声明,均可在 https://doi.org/10.1038/s41586-02 4-07578-8 获取。
1. Paulsen, B. et al. Autism genes converge on asynchronous development of shared neuron classes. Nature 602, 268–273 (2022).
2. Pizzo, L. et al. Rare variants in the genetic background modulate cognitive and developmental phenotypes in individuals carrying disease-associated variants. Genet. Med. 21, 816–825 (2019).
3. Ford, L. C. et al. A population-based human in vitro approach to quantify inter-individual variability in responses to chemical mixtures. Toxics 10, 441 (2022).
4. Germain, P.-L. & Testa, G. Taming human genetic variability: transcriptomic meta-analysis guides the experimental design and interpretation of iPSC-based disease modeling. Stem Cell Rep. 8, 1784–1796 (2017).
5. Tegtmeyer, M. et al. High-dimensional phenotyping to define the genetic basis of cellular morphology. Nat. Commun. 15, 347 (2024).
6. Cederquist, G. Y. et al. A multiplex human pluripotent stem cell platform defines molecular and functional subclasses of autism-related genes. Cell Stem Cell 27, 35–49 (2020).
7. Cuomo, A. S. E. et al. Single-cell RNA-sequencing of differentiating iPS cells reveals dynamic genetic effects on gene expression. Nat. Commun. 11, 810 (2020).
8. Jerber, J. et al. Population-scale single-cell RNA-seq profiling across dopaminergic neuron differentiation. Nat. Genet. 53, 304–312 (2021).
9. Limone, F. et al. Efficient generation of lower induced motor neurons by coupling Ngn2 expression with developmental cues. Cell Rep. https://doi.org/10.1016/j.celrep.2022.111896 (2023).
10. Mitchell, J. M. et al. Mapping genetic effects on cellular phenotypes with “cell villages”. Preprint at bioRxiv https://doi.org/10.1101/2020.06.29.174383 (2020).
11. Wells, M. F. et al. Natural variation in gene expression and viral susceptibility revealed by neural progenitor cell villages. Cell Stem Cell 30, 312–332 (2023).
12. Wozniak, J. R., Riley, E. P. & Charness, M. E. Clinical presentation, diagnosis, and management of fetal alcohol spectrum disorder. Lancet Neurol. 18, 760–770 (2018).
13. Bjørk, M.-H. et al. Association of prenatal exposure to antiseizure medication with risk of autism and intellectual disability. JAMA Neurol. 79, 672–681 (2022).
14. Christensen, J. et al. Prenatal valproate exposure and risk of autism spectrum disorders and childhood autism. JAMA 309, 1696–1703 (2013).
15. Neavin, D. R. et al. A village in a dish model system for population-scale hiPSC studies. Nat. Commun. 14, 3240 (2023).
16. Villa, C. E. et al. CHD8 haploinsufficiency links autism to transient alterations in excitatory and inhibitory trajectories. Cell Rep. 39, 110615 (2022).
17. Warren, C. R. & Cowan, C. A. Humanity in a dish: population genetics with iPSCs. Trends Cell Biol. 28, 46–57 (2018).
18. Velasco, S. et al. Individual brain organoids reproducibly form cell diversity of the human cerebral cortex. Nature 570, 523–527 (2019).
19. Kang, H. M. et al. Multiplexed droplet single-cell RNA-sequencing using natural genetic variation. Nat. Biotechnol. 36, 89–94 (2018).
20. Uzquiano, A. et al. Proper acquisition of cell class identity in organoids allows definition of fate specification programs of the human cerebral cortex. Cell 185, 3770–3788 (2022).
21. Dann, E., Henderson, N. C., Teichmann, S. A., Morgan, M. D. & Marioni, J. C. Differential abundance testing on single-cell data using k-nearest neighbor graphs. Nat. Biotechnol. 40, 245–253 (2022).
22. Cahill, K. M., Huo, Z., Tseng, G. C., Logan, R. W. & Seney, M. L. Improved identification of concordant and discordant gene expression signatures using an updated rank-rank hypergeometric overlap approach. Sci. Rep. 8, 9588 (2018).
23. Trevino, A. E. et al. Chromatin and gene-regulatory dynamics of the developing human cerebral cortex at single-cell resolution. Cell 184, 5053–5069 (2021).
24. Polioudakis, D. et al. A single-cell transcriptomic atlas of human neocortical development during mid-gestation. Neuron 103, 785–801 (2019).
25. Stuart, T. et al. Comprehensive integration of single-cell data. Cell 177, 1888–1902 (2019).
26. Cao, J. et al. The single-cell transcriptional landscape of mammalian organogenesis. Nature 566, 496–502 (2019).
27. Manno, G. L. et al. RNA velocity of single cells. Nature 560, 494–498 (2018).
28. Alfonso-Loeches, S. & Guerri, C. Molecular and behavioral aspects of the actions of alcohol on the adult and developing brain. Crit. Rev. Clin. Lab. Sci. 48, 19–47 (2011).
29. Arzua, T. et al. Modeling alcohol-induced neurotoxicity using human induced pluripotent stem cell-derived three-dimensional cerebral organoids. Transl. Psychiat. 10, 347 (2020).
30. Carpita, B. et al. Autism spectrum disorder and fetal alcohol spectrum disorder: a literature review. Brain Sci. 12, 792 (2022).
31. Charness, M. E. Fetal alcohol spectrum disorders: awareness to insight in just 50 years. Alcohol Res. 42, 05 (2022).
32. Eberhart, J. K. & Parnell, S. E. The genetics of fetal alcohol spectrum disorders. Alcohol Clin. Exp. Res. 40, 1154–1165 (2016).
33. Granato, A. & Dering, B. Alcohol and the developing brain: why neurons die and how survivors change. Int. J. Mol. Sci. 19, 2992 (2018).
34. Marguet, F. et al. Oligodendrocyte lineage is severely affected in human alcohol-exposed foetuses. Acta Neuropathol. Commun. 10, 74 (2022).
35. Streissguth, A. P. & Dehaene, P. Fetal alcohol syndrome in twins of alcoholic mothers: concordance of diagnosis and IQ. Am. J. Med. Genet. 47, 857–861 (1993).
36. Sulik, K. K., Johnston, M. C. & Webb, M. A. Fetal alcohol syndrome: embryogenesis in a mouse model. Science 214, 936–938 (1981).
37. Meng, Q. et al. Human forebrain organoids reveal connections between valproic acid exposure and autism risk. Transl. Psychiatry 12, 130 (2022).
出版者声明:施普林格·自然在已发布地图和机构隶属关系中的司法管辖权声明方面保持 中立。 Springer Nature或其许可方(例如学会或其他合作伙伴)根据与作者或其他权利持有人签订的 出版协议对本文享有专有权利;作者对本文接受稿版本的自我存档仅受此类出版协议条款和 适用法律的约束。
¶ 方法
伦理声明
所有涉及人类细胞系的实验均已获得哈佛大学IRB和ESCRO委员会的批 准。所有实验均按照相关指南和法规进行,并根据从原始细胞或组织的 捐赠者处获得的知情同意进行。
¶ 人类PSC培养
所有人类PSC细胞系均按照先前描述的方法进行培养1,18,20。简而言之, 使用MTESR1培养基(StemCell Technologies)、mTESR+培养基(Stem -Cell Technologies)或StemFlex培养基(Gibco),所有培养基中均添加 1%的青霉素-链霉素溶液(Corning),在预先涂有1% Geltrex(Gibco) 的细胞培养皿(Falcon)中培养干细胞,温度为37 °C,CO2浓度为5%。 所有PSC细胞系均保持在传代50以下,并经检测为阴性,无支原体污染 (使用MycoAlert PLUS支原体检测试剂盒,Lonza进行检测)。
¶ PSC细胞系的特征描述
此处使用的PSC细胞系包括:H1(男性,人类胚胎干细胞(hES)), Mito210(男性,诱导多能干细胞(iPSCs);无神经系统疾病家族史的 对照),PGP1(男性,iPSCs,对照),CW50037(女性,iPSCs,无 神经系统疾病家族史的对照),GM08330(男性,iPSCs,临床上未受 影响但有相关精神疾病家族史)和11a(男性,iPSCs,对照)。
Mito210 iPSC细胞系由B. Cohen提供;PGP1 iPSC细胞系38由G. Church 提供;GM08330 iPSC细胞系39由M. Talkowski提供,最初来源于从Corie ll医学研究所获得的成纤维细胞40。CW50037 iPSC细胞系来自加州再生 医学研究所的iPSC收藏。H1 hES细胞系(也称为WA01)41是从WiCell 购买的;11a iPSC细胞系42是从哈佛干细胞研究所获得的。
所有细胞系的认证如下:PGP1 iPSC细胞系通过短串联重复分析进行 认证(由TRIPath于2018年执行)20。Mito210 iPSC细胞系通过基因分型 分析(使用Fluidigm FPV5芯片)进行认证,由Broad Institute基因组平 台执行20。H1和GM08330细胞系通过短串联重复分析进行认证(由WiC ell于2021年执行)。CW50037 iPSC细胞系使用SNP基因分型(Illumina 的Illumina Global Screening Array (GSA);在Broad Institute的基因组平台 处理)进行细胞系识别和染色体异常检测。GM08330亲本细胞系在20号 染色体长臂(q)上有一个先前报道的间质重复(ref. 1);其他所有细胞 系的核型均正常。
¶ 皮层类器官分化
背侧模式的PSC-Chimeroids是根据先前描述的协议1,18,20生成的。简而言 之,在第0天,无饲养层培养的人iPSCs或hES细胞,达到75-85%汇合度 后,用Accutase (Gibco)酶解成单细胞,并在超低细胞粘附的96孔板中以 每孔9,000个细胞的密度重新聚集,使用V形底锥形孔(sBio PrimeSurfac e板;住友电木)在之前维持的相同多能细胞培养基中进行培养。在第1 天,80 μl的培养基被替换为皮质分化培养基(CDM)I,其中含有Glasg ow-MEM (Gibco)、20% Knockout Serum Replacement (Gibco)、0.1 mM最 低必需培养基非必需氨基酸(MEM-NEAA)(Gibco)、1 mM丙酮酸盐(G ibco)、0.1 mM 2-巯基乙醇(Gibco),以及1%青霉素-链霉素溶液(Corning) 。从第0天到第6天,ROCK抑制剂Y-27632 (Millipore)以20 μM的终浓度 加入培养基中。模式化小分子WNT抑制剂IWR1 (Calbiochem)和TGFβ抑 制剂SB431542 (Stem Cell Technologies)从第0天到第15-18天分别以3 μM 和5 μM的浓度加入。
从第15天到第18天,图案化的胚状体在轨道搅拌(Thermo Fisher Scie ntific,CO2 -耐受轨道摇床)下,在超低附着力培养皿(Corning)中使 用CDM II培养基培养,其中包含DMEM/F12培养基(Gibco)、2 mM Gl utaMax(Gibco)、1% N2(Gibco)、1%化学定义的脂质浓缩物(Gibc o)、0.25 μg ml−1 fungizone(Gibco),以及1%的青霉素-链霉素溶液( Corning)。在第35天,细胞聚集体被转移到旋转瓶生物反应器(Cornin g)中,并在CDM III中维持(CDM II补充10%胎牛血清(GE-Healthcare )、5 μg ml−1肝素(Sigma-Aldrich)和1% Matrigel(Corning))。从第 70天开始,类器官在CDM IV中培养(CDM III补充B27补充剂(Gibco )和2% Matrigel)。请注意,对于PSC-Chimeroids,起始点是解离的iPS Cs或ES细胞用于胚状体的创建,依据先前报道的类器官协议1,18,20。
¶ Chimeroid 聚集步骤
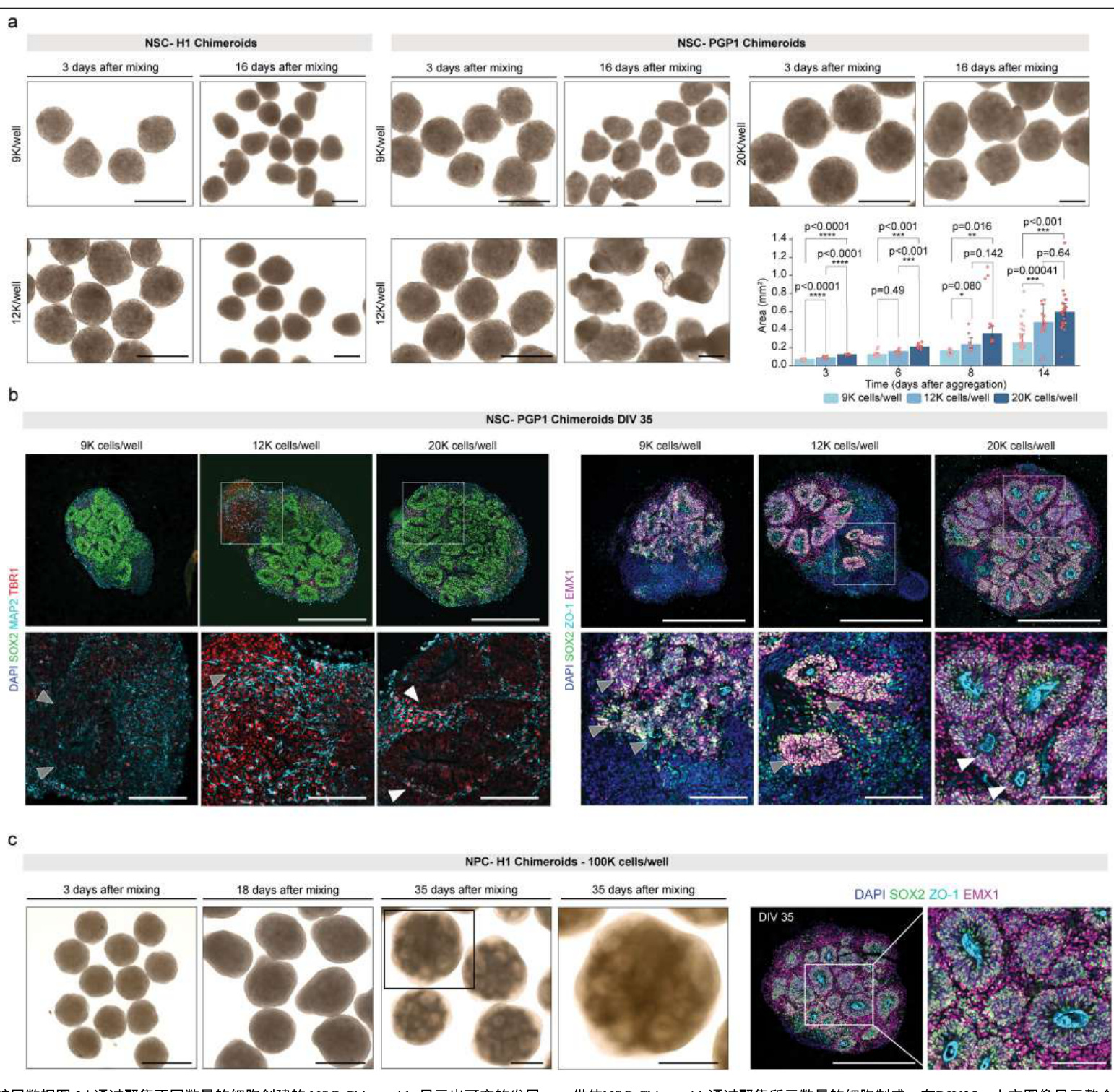
我们在NSC-Chimeroid生成过程中测试了9,000、12,000和15,000个细胞 的聚集步骤。由少于15,000个细胞聚集而成的Chimeroid仍然非常小且大 小不均匀,IHC分析显示其向皮质命运的分化很少(扩展数据图 2)。 因此,我们将每孔的播种细胞数增加到15,000–18,000(数据未显示) 和20,000个细胞。我们观察到每孔15,000、18,000、20,000和100,000个 细胞成功生成了遵循我们既定类器官协议18的正确发育轨迹的Chimeroid (扩展数据图 2)。对于NPC-Chimeroid,我们将细胞数量增加到50,000 ,因为在这个阶段的一部分细胞不再对类器官的进一步发育做出贡献, 并且在这个阶段大约20%的类器官细胞是后有丝分裂的20;这些后有丝 分裂的细胞没有能力重新进入细胞周期,并且在聚集后不太可能对Chi meroid的发育做出贡献。
¶ 多供体皮质嵌合体生成
我们使用了以下供体组合:混合1(4条:CW,H1,Mito210,PGP1) ,混合2(5条:CW,H1,Mito210,PGP1,11a),混合3(4条:GM ,H1,Mito210,PGP1),混合4(5条:GM,H1,Mito210,PGP1,1 1a),混合5(4条:CW,H1,PGP1,11a)和混合6(GM,CW,Mit o210,H1,PGP1,11a)。每个实验中包含的组合详情在补充表 1中提 供。
Chimeroid 生成基于一种重聚过程,该过程使用源自背侧模式端脑类 器官(皮层类器官)的假定 NSC 或 NPC,在不同发育阶段解离;
NSCChimeroids 和 NPC-Chimeroids 分别来源于 DIV15–18 和 DIV23–25 的 皮层类器官。 由于 NSC-Chimeroid 协议在我们测试的大多数细胞系中效果良好,我 们认为在不需要包含具有极端增殖或分化偏向的 PSC 细胞系的情况下 ,它是首选协议。我们开发了 NPC-Chimeroid 协议,以便结合具有非常 不同增殖或分化特性的 PSC 细胞系。
在所需的发育阶段,每位供体的15-30个皮质类器官池被酶解解离成 单细胞悬液,使用Worthington Papain Dissociation System试剂盒(Worth ington Biochemical)。该方案基于先前发表的协议18,并进行了调整以 生成Chimeroids。具体而言,通过明场显微镜检查皮质类器官以确认其 形态正确,并收集到低结合1.5 ml管(Eppendorf)中。在酶解解离之前 ,去除多余的培养基。将加热的木瓜蛋白酶溶液(37 °C,500 μl)加入 管中,使用P1000移液器反复吹打10次,然后在37 °C下在轨道摇床上以 90 rpm振荡孵育25 分钟。然后使用P1000移液器进一步吹打组织块,随 后在37 °C下再孵育5 分钟。所得溶液被转移到含有1 ml Earle平衡盐溶液 和600 μl卵粘蛋白抑制剂的15 ml管(Falcon)中。溶液,然后在 300g下离心 5 分钟。弃去上清液,将解离的细胞重悬于 含有 20 μM ROCK 抑制剂 Y27632 (Millipore) 的 1 ml CDM1 中。为去除 聚集体并制备单细胞溶液,使用 35 μm 细胞过滤管 (Corning) 过滤解离 的细胞,然后使用 Countess II 自动血细胞计数器 (Thermo Fisher Scientif ic) 进行计数。来自不同供体的细胞悬液按相同比例混合,每孔重新聚 集 18,000–20,000 个细胞(调整以确保每个供体至少 1,000 个细胞)在 超低细胞粘附的 96 孔板中,带有 V 型底锥形孔 (sBio PrimeSurface plate ; Sumitomo Bakelite)。第二天,将培养基更换为每孔 100 μl 的 CDM2。 然后,在混合后 2 天,将重新聚集的胚状体在超低附着力培养皿 (Corni ng) 中传代,并在轨道振荡 (Thermo Fisher Scientific, CO2 耐受轨道振荡 器) 下培养。Chimeroids 的后续培养基更换根据我们已发表的协议18进行 ,在 DIV35 更换为 CDM3 培养基,然后在 DIV70 更换为 CDM4。
¶ 乙醇和VPA处理
多供体和单供体嵌合体从第45天到第75天接受了乙醇或VPA的处理。选 择这个时间窗口是因为它针对广泛的细胞类型,包括所有潜在的祖细胞 类型以及所有起源于皮层的神经元细胞类型,包括aRG细胞、IPs、oRG 细胞和各种谷氨酸能锥体神经元的子集,如CFuPN和CPN20。为了模拟 怀孕期间典型的酒精暴露的生理波动,乙醇(E7023-6,Millipore-Sigma )以50 μM的浓度直接添加到培养基中,每2 天一次,并在施用后用Para film M(Bemis)密封培养板24 小时以避免蒸发29。VPA(P4543-25G, Millipore-Sigma)以0.7 mM的浓度(一个临床相关的浓度)添加到培养 基中,并且每周更换两次培养基。两种处理均在DIV75时停止,并在MI V3时对类器官进行分析。
¶ 样品的固定和处理以进行冷冻切片
嵌合体在4 °C的12孔板(Falcon)中用4%多聚甲醛(电子显微镜服务) 固定过夜,然后用1×磷酸盐缓冲盐水(PBS)(Gibco)洗涤三次,并 在4 °C的PBS中用30%蔗糖溶液(Sigma-Aldrich)进行低温保护(过夜 )。然后将样品嵌入牛明胶中。含有10%牛明胶(Sigma-Aldrich)和7.5 %蔗糖(Sigma-Aldrich)的明胶溶液在37 °C预热15 分钟。将样品中的30 %蔗糖溶液移除并更换为预热的明胶,样品在37 °C孵育15 分钟。同时 ,塑料模具涂上一层2 毫米的温暖明胶溶液,并在室温下聚合。将嵌合 体转移到预处理的塑料模具中,加入1 毫升温暖的明胶溶液覆盖嵌合体 。在室温下聚合3 分钟后,嵌合体在4 °C预冷15–20 分钟。最后,将模 具在含有100%乙醇和干冰的冷浴中冷冻2–3 分钟,并无限期存储在−80 °C。
¶ IHC 分析
对于IHC,使用冷冻切片机(Leica)切割14至20-μm厚的切片。 冷冻切 片在室温下稳定5 分钟,并在PBS中用10%驴血清(Sigma-Aldrich) + 0. 3% Triton X-100(Sigma-Aldrich)封闭。将一抗(补充表 2)稀释在封 闭液中并孵育过夜。经过四次PBS洗涤后,冷冻切片在室温下用稀释在 PBS中的二抗(1:1,000;补充表 2)孵育1 小时,在室温下用PBS洗涤四 次,并用DAPI(1:10,000在PBS + 0.1% Tween-20中)染色15 分钟以可视 化细胞核。
¶ 显微镜学
免疫荧光图像是使用 Zeiss Axio Imager.Z2 或 Zeiss LSM900 共聚焦系统 获取的。在 Axio Imager.Z2 上的图像是使用 ×20 倍物镜(像素大小为 0. 325 μm)并使用 Apotome 光学切片功能获取的。使用 Zen Blue 软件进 行拼接和 Apotome 去卷积处理后导出图像。对于 LSM900,使用 z 堆栈 以 3 μm 步长获取图像,使用 ×20 倍物镜(像素大小为 0.62 μm),然 后使用 Zen Blue 软件进行拼接。进一步的处理,如 z 投影、通道合并和 添加比例尺,在 Fiji43 中进行。图像的亮度和对比度进行了调整;所有 调整均应用于整个图像。
¶ 电生理分析
MEA电生理记录在MIV4的混合2 NSC-Chimeroids中进行,此时该类类 器官模型通常具有电活动1。使用3Brain的Accura 3D CMOS-HD-MEA系 统(与Accura 3D芯片结合的BioCAM DupleX系统)记录分布在类器官 大面积上的单元的电信号44。3D芯片包含4,096个穿透μ针状电极,高度 为90 μm,排列在64 × 64的网格中,测量为3.8 × 3.8 mm,采样率为20 kH z,分辨率为12位。在分析前14-21天将类器官更换为BrainPhys培养基1, 并在37 °C的迷你培养箱中充满Carbogen进行完整类器官的急性记录。使 用BrainWave v.5记录自发活动15 分钟。为了测试测量的电活动是否为 突触活动,在记录结束时使用D-AP5(150 μM)和DNQX(30 μM)阻 断NMDA和AMPA受体活动。使用BrainWave软件检测尖峰,使用Brain Wave中实现的精确定时尖峰检测算法在原始通道轨迹上应用检测阈值 为8 s.d.
¶ 脑类器官的解离和单细胞RNA测序
类器官的解离方法如前所述18,但MIV1类器官除外,其解离使用低结合 1.5 ml管(Eppendorf),具体方法在“多供体皮质嵌合体生成”部分中 描述。解离后的细胞重悬于100 μl PBS中,并通过35 μm细胞过滤管(C orning)过滤以避免聚集,然后使用Cellometer K2仪器(Nexcelom)或 Countess II自动血细胞计数器(Thermo Fisher Scientific)进行计数。单 细胞悬液被加载到Chromium Next GEM Chip G(10x Genomics, 1000120 )中,并通过Chromium控制器运行以生成乳液中的单细胞凝胶珠。scR NA-seq文库使用Chromium Single Cell 3’ Library and Gel Bead Kit v3.1 (10x Genomics, 1000268)制备。当嵌合体由来自多个遗传背景的细胞 组成时,可以利用这种遗传变异来识别含有来自两个不同细胞系的细胞 的双细胞。这使我们能够容忍更高的双细胞率,并在每个通道中加载约 20,000–30,000个细胞。生成的文库根据摩尔浓度进行合并,并在NextS eq 500或NovaSeq 6000仪器(Illumina)上进行测序,read 1为28个碱基 ,read 2为55个碱基,index read 1为8个碱基。如有必要,在第一次测序 后,我们根据每个文库中的实际细胞数重新合并文库,并重新测序,目 标是为每个样本生成相同数量的每细胞读数(目标读深为每细胞20,000 个读数)。
¶ scRNA-seq 数据处理
所有 scRNA-seq 数据均使用 10x Genomics Cell Ranger (v.7.1.0)45 处理。 使用 mkfastq 命令和默认参数将原始 BCL 文件转换为 FASTQ 文件。对 生成的 FASTQ 文件使用 CellRanger 的 count 功能,以生成每个类器官 的细胞基因计数矩阵,使用预构建的 GRCh38 人类参考。基因组提供在10x Genomics下载页面(https://support.10xgenomics.com/si ngle-cell-gene-expression/software/downloads/latest; GENCODE v32/Ensem bl 98)。每个样本的expect-cells标志手动设置,值范围从10,000到30,00 0。所有其他参数均设置为默认值。使用R(v.4.2.2)将CellRanger的filte red_feature_bc_matrix导入Seurat(v.4.3.0)25进行下游分析。
¶ 基因解复用
对于基于scRNA-seq数据的遗传去复用,应用了demuxlet19,使用默认设 置,处理由CellRanger生成的比对结果。通过对每个供体细胞系进行全 基因组测序生成的参考变异调用格式(VCF)文件,使用bcftools46进行 处理,以将参考VCF标准化为双等位基因变异,并去除不通过和单态变 异。在下游分析之前,从Seurat对象中移除了demuxlet确定具有不明确 遗传来源或可能是异质双重体的液滴。
为了确定在不同时间点和不同混合物中的PSC-、NSC-和NPC-Chimero ids中每个供体的比例丰度,Census-seq被应用于低覆盖率WGS数据11, 使用Drop-seq软件包中的Census-seq函数,采用默认参数,遵循GitHub 上提供的Census-seq计算协议(https://github.com/broadinstitute/Drop-seq ),并使用上述提到的每个供体的相同参考VCF文件。
¶ 细胞轮廓质量过滤、标准化、聚类,注释和集成
对于每个Chimeroid,去除细胞谱中表达基因少于200个、唯一分子标识 符(UMIs)少于500个、多于20,000个UMIs或线粒体RNA超过15%的细 胞。这些阈值最初是为了保留所有细胞类型以及可能受到乙醇或VPA暴 露不利影响的细胞;然而,在最终数据集中,只有0.15%的分析细胞表 达少于500个基因,少于0.43%的分析细胞的线粒体RNA含量超过10%。 在每个类器官的过滤计数矩阵上运行Seurat的SCTransform函数,回归掉 线粒体RNA的百分比。在SCTransform找到的可变特征上进行PCA。使 用Seurat的FindNeighbors函数构建一个k-最近邻图(k = 20),使用前30 个主成分(PCs),然后使用FindClusters函数进行Louvain聚类,分辨率 设置在0.2到1.0之间。
来自多个Chimeroids的单细胞表达数据被合并成几个更大的联合数据 集,并根据协议和处理进行如下处理:混合5的PSC-Chimeroids (n = 2; 1 5,612个细胞) 使用Seurat的merge函数合并,然后使用SCTransform进行 联合重新标准化;混合3和混合4的NSC-和NPC-Chimeroids (n = 6; 52,771 个细胞) 根据Seurat的集成小册子进行集成,具体来说,使用SelectIntegr ationFeatures(nfeatures参数设置为3000)、PrepSCTIntegration、FindInt egrationAnchors和IntegrateData函数;混合1和混合2的对照、乙醇处理和 VPA处理的多供体NSC-Chimeroids以及对照、乙醇处理和VPA处理的单 供体NSC-Chimeroids (n = 93; 420,288个细胞) 通过两步过程合并:多供 体NSC-Chimeroids使用Seurat的merge函数在其原始计数矩阵上合并,使 用Seurat的NormalizeData、FindVariableFeatures(nfeatures设置为3000) 和ScaleData函数进行联合重新标准化和缩放,并在联合表达矩阵上执行 PCA,然后使用与单个数据集相同的方法构建k-最近邻图和聚类。具有 异常低UMI计数的簇被标记为低质量细胞并移除(20,581个细胞)。单 供体NSC-Chimeroids以类似方式合并、处理和过滤(移除4,133个细胞) 。然后使用Seurat的merge函数合并联合多供体和联合单供体NSC-Chime roids数据集的原始计数矩阵。
来自多个Chimeroids的单细胞表达数据被合并成几个更大的联合数据 集,并根据协议和处理进行如下处理:混合5的PSC-Chimeroids (n = 2; 1 5,612个细胞) 使用Seurat的merge函数合并,然后使用SCTransform进行 联合重新标准化;混合3和混合4的NSC-和NPC-Chimeroids (n = 6; 52,771 个细胞) 根据Seurat的集成小册子进行集成,具体来说,使用SelectIntegr ationFeatures(nfeatures参数设置为3000)、PrepSCTIntegration、FindInt egrationAnchors和IntegrateData函数;混合1和混合2的对照、乙醇处理和 VPA处理的多供体NSC-Chimeroids以及对照、乙醇处理和VPA处理的单 供体NSC-Chimeroids (n = 93; 420,288个细胞) 通过两步过程合并:多供 体NSC-Chimeroids使用Seurat的merge函数在其原始计数矩阵上合并,使 用Seurat的NormalizeData、FindVariableFeatures(nfeatures设置为3000) 和ScaleData函数进行联合重新标准化和缩放,并在联合表达矩阵上执行 PCA,然后使用与单个数据集相同的方法构建k-最近邻图和聚类。具有 异常低UMI计数的簇被标记为低质量细胞并移除(20,581个细胞)。单 供体NSC-Chimeroids以类似方式合并、处理和过滤(移除4,133个细胞) 。然后使用Seurat的merge函数合并联合多供体和联合单供体NSC-Chime roids数据集的原始计数矩阵。 函数并重新归一化、重新缩放和重新聚类使用相同的方法。
对每个组合的scRNA-seq数据集进行了PCA分析,并通过统一流形近 似和投影(UMAP)嵌入细胞特征(前30个主成分)以进行可视化,然 后进行了最后一轮k最近邻图构建和聚类。在每个数据集中,每个聚类 根据以下组合手动注释为细胞类型标签:(1)经典标记基因表达;(2 )通过Seurat的FindMarkers函数确定的每个聚类内上调基因列表;(3 )使用Seurat的FindTransferAnchors和MapQuery函数从我们先前发布的 Velasco协议类器官发育参考图谱的表达特征中自动标签转移。在每个 合并或集成的数据集中,基于联合聚类标签进一步细化细胞类型标签( 补充表 9–12)。细胞特征(前30个主成分)通过UMAP嵌入。具有异 常低UMI计数的聚类被标记为低质量细胞,并从下游分析中移除。最初 只能描述为循环祖细胞的聚类通过仅选择这些聚类中的细胞并重新运行 PCA、k最近邻图构建和聚类进行子聚类;使用HOPX、EOMES和SOX2 的表达来识别这些子聚类为aRG细胞、oRG细胞或IPs。
¶ 先前发表的人类胎儿数据分析---三个人类胎儿皮层的先前已发表数据集被使用用于比较。
首先,从CoDEx查看器(http://solo.bmap.ucla.edu/shiny/webapp/)下 载了一个先前的Drop-seq数据集24,该数据集包含来自人类胎儿大脑皮 层样本的33,986个细胞,孕周为17和18周,并附有相关的元数据(包括 每个细胞的细胞类型分配)。使用原作者分配的细胞类型标签计算每对 样本之间的Aitchison距离。
其次,从https://github.com/GreenleafLab/brai nchromatin/blob/main/links.txt下载了一个先前的scRNA-seq数据集23,该 数据集包含来自孕周为16、20、21和24周的人类胎儿大脑皮层样本的57 ,868个单细胞转录组,并附有相关的元数据(包括细胞类型分配)。使 用原作者分配的细胞类型标签计算每对样本之间的Aitchison距离。
第三,我们之前的snRNA-seq数据集20来自4位供体的胎儿皮层样本( 受孕后14、15、16和18周),我们将其子集化以仅包括与MIV3类器官 或Chimeroids相关(或具有密切类似物)的细胞类型(aRG, oRG, IP, CF uPN, CPN, 第IV层, CGE中间神经元, 胶质前体和少突胶质细胞前体细胞 )。这产生了52,658个细胞,用于测量数据集中胎儿样本之间的Aitchiso n距离,以及用于关联胎儿数据和Chimeroids中不同细胞类型的标记基因 表达,并将Chimeroid细胞类型标签映射到胎儿细胞上(图 3)。为了使 用参考标签转移将胎儿细胞类型标签映射到Chimeroids上,该数据集进 一步子集化为每种细胞类型最多5,000个细胞,并去除第IV层细胞。
¶ 轨迹分析
使用velocyto Python软件包计算每个未处理的混合1/混合2 MD NSC-Chi meroid的剪接和未剪接计数矩阵。这些剪接/未剪接矩阵被合并,并使用 Python软件包scVelo计算每个细胞中每个基因的RNA速度,并投影到组 合MD/SD Chimeroid Seurat对象的UMAP嵌入中。对于每个未处理的SD NSC-Chimeroid重复相同的过程。使用scVelo的pl.velocity_embedding_gri d函数可视化速度。
伪时间分别为每个数据集计算,包括MIV3 MD NSC-Chimeroids、MI V3 SD NSC-Chimeroids、MIV3 NPC-Chimeroids和MIV3参考图谱类器官 (仅包含源自PGP1或Mito210细胞系的MIV3类器官),使用Monocle{v *}3(v.1.3.4)在R中以aRG细胞为根细胞。对于每个数据集,伪时间值被缩放到最小值为0,最大值为1。使用ggplot2的geom_density_ridges函 数可视化每个数据集中每种细胞类型和供体在伪时间上的分布。
¶ 糖酵解特征分析
来自MSigDB的Hallmark集合的糖酵解基因集,其中包含200个基因,用 于计算从这些数据集中抽取的20,000个细胞随机子集的模块分数:MIV3 MD NSC-Chimeroids、SD NSC-Chimeroids和NPC-Chimeroids,以及参 考图谱类器官样本(子集仅包括来自PGP1或Mito210细胞系的MIV3类 器官),使用Seurat的AddModuleScore函数。为了测试不同的类器官和 Chimeroid协议是否在每种主要细胞类型(aRG, oRG, IP, PN, CFuPN, CP N和中间神经元)中显示不同的糖酵解表达模式,糖酵解模块分数在每 个类器官(对于参考图谱类器官)和每个Chimeroid的每个供体中每种 细胞类型的所有细胞中进行平均。对于每种细胞类型,使用混合效应线 性模型比较每对数据集之间的糖酵解模块分数,以供体来源作为随机效 应。使用R中的基础anova函数比较包含和不包含“协议”作为协变量的 模型。
¶ 可重复性及与胎儿组织和皮层的比较类器官
Aitchison距离定义为每个样本中中心对数比转换的细胞类型丰度之间的 欧几里得距离,使用R包robCompositions(v.2.4.1)的aDist函数计算每 个可能的样本对之间的距离(一个样本是来自同一供体在类器官或Chi meroid中的所有细胞)。Aitchison距离测量成分数据中的成对差异,考 虑到任何给定样本中的总细胞类型丰度必须总和为100%(因此,一种 细胞类型的扩展必然导致其他细胞类型丰度的减少)。使用Wilcoxon秩 和检验比较每个协议内重复之间的Aitchison距离。
为了比较个体匹配供体系内不同协议的变异性,特别比较了PGP1和 Mito210样本的批内和批间距离(因为这些是唯一存在于所有四个相关 协议中的供体系:MD-NSC-、SD-NSC-和MD-NPC-Chimeroids,以及参 考图谱类器官)。这些成对的Aitchison距离进一步按是否代表两个PGP 1样本、两个Mito210样本或一个PGP1和一个Mito210样本进行分组。
Seurat 的 FindTransferAnchors 和 MapQuery 函数按照 Seurat 4.3 标签 转移教程中的描述,用于将标签从胎儿皮层样本和皮层类器官转移到 C himeroids 和单供体 Chimeroids(反之亦然)。
Seurat 的 FindMarkers 函数在没有折叠变化或显著性阈值的情况下被 用来独立创建未处理的 NSC-Chimeroids、胎儿皮质组织或参考图皮质类 器官中每种细胞类型的标记基因列表,按显著性排序,显著性通过 Wilc oxon 秩和检验计算,列表顶部是某种细胞类型中显著上调的基因,底 部是显著下调的基因。使用 RRHO2 R 包(v.1.0)中的改进的秩–秩超 几何重叠测试来比较每种 NSC-Chimeroid 细胞类型的标记基因列表与每 种胎儿和类器官标记列表。
Seurat 的 FindMarkers 函数在没有折叠变化或显著性阈值的情况下被 用来独立创建未处理的 NSC-Chimeroids、胎儿皮质组织或参考图皮质类 器官中每种细胞类型的标记基因列表,按显著性排序,显著性通过 Wilc oxon 秩和检验计算,列表顶部是某种细胞类型中显著上调的基因,底 部是显著下调的基因。使用 RRHO2 R 包(v.1.0)中的改进的秩–秩超 几何重叠测试来比较每种 NSC-Chimeroid 细胞类型的标记基因列表与每 种胎儿和类器官标记列表。
对于19个典型细胞类型标记基因中的每一个,计算了每种主要细胞类 型(aRG、oRG、IP、CFuPN和CPN)在5个数据集(混合1/混合2 NSCChimeroids、NPC-Chimeroids、单供体Chimeroids、参考图谱类器官和 内源性胎儿皮层)中每个细胞的平均缩放表达,以比较可比细胞类型之 间的数据集中的标记基因表达。计算了这些表达值在内源性胎儿组织与 相应细胞类型之间的Pearson相关系数。 在每个类器官和嵌合体数据集中,以及在参考图谱类器官和每个嵌合体 数据集之间。
EdgeR (v.3.40.2)47–49 用于测试 MD-NSC-Chimeroids 和 SD-NSC-Chime roids、MD-NPC-Chimeroids 和参考图谱类器官之间的共同供体系内细胞 类型丰度的差异。使用 MD-NSC-Chimeroids 作为基线,报告每个供体 系和协议的差异。
¶ 细胞类型丰度、供体丰度和邻域治疗条件之间的变化
使用负二项混合效应模型来测试对照Chimeroids与VPA或乙醇处理的Chi meroids之间的细胞类型丰度差异。具体而言,应用lme4(v.1.1.33)的g lmer.nb函数到每个Chimeroid内每个供体的细胞类型计数矩阵,使用每 个供体和Chimeroid的细胞总数的对数作为偏移量,Chimeroid分化批次 和供体混合(混合1或混合2)作为固定效应,并将供体身份(以考虑供 体之间细胞类型丰度的总体差异和在重复中供体内的相似性)和个体Ch imeroid(以考虑在共享类器官内供体之间细胞类型丰度的相似性)作为 随机效应。因此,使用了Cell Type ~ Treatment + offset(log(LibrarySize)) +Batch + Mix + (1|Chimeroid) + (1|Donor)的模型公式。通过使用R stats包 v.4.2.2中的anova函数,将此模型与没有处理固定效应但其他方面相同的 模型进行比较,计算处理对细胞类型丰度的影响显著性。基于所得的P 值计算Benjamini–Hochberg FDR调整的P值。
EdgeR (v.3.40.2)47–49 被用于测试供体丰度在 VPA 或乙醇处理下的变 化。具体来说,将每个 Chimeroid 中每个供体的细胞计数矩阵通过函数 calcNormFactors(方法设置为 TMM)、estimateDisp(趋势设置为 none )、glmQLFit(abundance.trend 设置为 FALSE)和 glmQLFTest,使用 设计公式将 Chimeroid 分化批次和供体混合作为附加协变量。显著性以 FDR 报告,使用 Benjamini–Hochberg 方法计算。
miloR R 包 (v.1.6.0)21 被用于评估重叠细胞邻域内的差异丰度,邻域大 小范围为每个 50–200 个细胞。对于每种处理(VPA 和乙醇),表达 矩阵被子集化以仅包括来自对照和处理的 Chimeroids 中变异性最高的 3, 000 个基因,在该子矩阵上执行 PCA,并将结果传递到 Milo 对象中进 行进一步分析。miloR 的 buildGraph 函数使用 k = 30 和 d = 30 运行,随 后是 makeNhoods 和 calcNhoodDistance。使用 testNhoods 进行差异丰度 测试,在每个邻域内对处理和对照细胞进行测试,设计为 ~Mix + Donor + Treatment。显著性报告为由 miloR 基于差异丰度 P 值和邻域之间的关 系计算的空间 FDR 值。相同的方法用于比较未经处理的 MD NSC-Chim eroids 和未经处理的 SD NSC-Chimeroids,省略了“Mix”协变量,因为 SD Chimeroids 不是供体的混合物。
¶ 伪批量分析和差异表达分析
为了总结乙醇和VPA处理的嵌合体中的基因表达变化,我们通过PCA降 维后确定样本之间的距离。对于每组伪批量表达谱(全局,aRG,oRG ,IP,CFuPN和PN),我们进行了PCA以识别主要的变异轴,然后比较 了来自同一供体的对照和VPA处理的嵌合体之间的欧几里得距离,该距 离是在所有PC上计算的。伪批量谱既可以在全局生成(其中伪批量谱 包括来自一个嵌合体中一个供体的所有细胞的UMI),也可以在逐细胞 类型的基础上生成(其中伪批量谱包括来自一个嵌合体中一个供体的某 一类型的所有细胞的UMI),通过汇总UMI。每个基因的计数,并在适当的情况下,将表达值计算为每个伪批量配置 文件中的百万计数的对数转换值。包含少于20个细胞的伪批量配置文件 从下游分析中移除。
对全局和个体细胞类型表达谱的全局伪合并表达矩阵中高度可变基因 (定义为平均表达加上方差与平均表达比的对数大于1的基因)进行了P CA。使用stats R包的dist()函数计算每个PCA中所有PC(每种情况下PC 的数量等于通过质量和异常值过滤的伪合并样本数量)之间的伪合并谱 的欧几里得距离。进行ANOVA以评估对照和处理样本之间的距离在五 个供体系之间是否显著不同。当ANOVA得出P值小于0.05时,进行事后 Tukey检验以进行供体之间的成对比较。
对于每个供体,通过将DESeq2应用于伪汇总表达矩阵,并使用分化 批次作为附加协变量,进行未处理和VPA处理的多供体NSC-Chimeroids 之间以及未处理和VPA处理的单供体NSC-Chimeroids之间的差异表达分 析(DEA)。在多供体分析中,仅在一个供体中发现显著差异表达的基 因(FDR调整后P < 0.05且log2转换的倍数变化绝对值为>0.5)被认为是 该供体“独有”的。这些每个供体的独有基因被用作GSEA中相应单供 体DEA结果的自定义基因集。对于每个供体,基因根据未处理和VPA处 理的单供体NSC-Chimeroids之间倍数变化的绝对值进行排名,并使用Ko lmogrov-Smirnov检验来测试供体特异的独有基因是否比随机基因具有更 高的排名。同样的分析也在另一个方向上进行:在单供体DEA中找到每 个供体的独有基因,并用作多供体DEA中GSEA的基因集。
为了评估未处理的混合物1/混合物2 MD NSC-Chimeroids中供体之间 的转录差异,使用DESeq2对这些供体的全局伪批量化谱进行分析,在 每对供体(CW-H1、CW-Mito210、CW-PGP1、CW-11a、H1-Mito210 、H1-PGP1、H1-11a、Mito210-PGP1、Mito210-11a和PGP1-11a)之间 进行DEA。在未处理的SD NSC-Chimeroids中,对相同的供体对进行了 等效的DEA。为了确定供体之间的转录差异是否在两个协议中保持一致 ,计算了每个协议中发现的对数2转化的折叠变化的Pearson’s r相关系 数。此外,进行了Fisher精确检验,以评估在每个协议中发现显著差异 表达的基因(调整后的P < 0.05和对数2转化的折叠变化的绝对值为>0.5 )是否有显著重叠。
同样,为了评估SD和MD NSC-Chimeroids之间的转录差异,DESeq2 被应用于每个细胞系的伪批量化谱,既是全局的也是针对每个主要细胞 类型(aRG, oRG, IP, CFuPN和CPN),以“protocol”(SD对比MD) 作为测试变量,并将批次作为附加协变量。此外,使用所有细胞系进行 类似的测试,包括起源供体的附加协变量。
我们没有发现任何基因在SD与MD NSC-Chimeroids中被VPA显著地以 相反方向失调;只有极小比例的基因(0.14–1.23%)在一个方案中被V PA显著扰动,但在另一个方案中具有相反符号的(不显著)log2转化的 倍数变化。尽管这些基因的数量太少,无法支持详细分析(中位数,每 个供体24个),但这些基因在未处理的单供体和多供体NSC-Chimeroids 之间差异表达的基因中富集。 嵌合体的空间转录组分析以绘制个体供体图谱。
为了绘制嵌合体中个体供体的空间组织,我们使用了Curio Seeker 3 × 3 试剂盒,按照制造商的协议(CB-000470-UG v2, Curio Biosciences)。 一个单独的MIV2嵌合体在含有2 U ml−1 RNase抑制剂(N8080119, Therm o Fisher Scientific)的PBS(100-10-023, Gibco)中洗涤两次。随后,嵌 合体在组织冷冻介质(72592-B, Electron Microscopy Sciences)中孵育1 分钟,并嵌入BEEM平嵌模具(70904-12, Electron Microscopy Sciences )中。模具被转移到干冰上20 分钟,并储存在−80 °C。切片前,模具和 CryoCube(Curio Seeker 3 × 3试剂盒的一部分)在冷冻切片机中平衡至− 18 °C 20 分钟。使用冷冻切片机(CM1950, Leica Biosystems)以10 μm 的厚度获得组织切片,并用刷子拉直。切片被小心地安装在Curio Seeke r 3 × 3 mm瓷砖上,并通过将手指放在瓷砖下方来融化。一个30-μm厚的 CryoCube切片被放置在组织切片上方,以类似方式融化并在−80 °C下冷 冻最多3 天。两个嵌合体切片,相隔20 μm,被安装在两个单独的瓷砖 上。然后根据试剂盒的建议(CB-000470-UG v2, Curio Biosciences)处 理样品。在标签化步骤中,使用了1,200 pM DNA,按照推荐的协议在N extera XT Library Prep Kit(FC-131-1024, Illumina)中进行。
文库在NextSeq 1000系统上进行合并和测序,读取长度如下:读取1 ,50 bp;索引1,8 bp;索引2,8 bp;读取2,50 bp。测序文件通过Curi o Seeker管道v.2.0.0处理(在线工具可在Curio Bioscience平台上获取:htt ps://curiobioscience.com/bioinformatics-pipeline/)进行基因表达分析。
比对后的读取被划分到每个空间索引珠子的单独 BAM 文件中。对每 个 BAM 文件应用 Census-seq,使用上述描述的参考 VCF 文件,以确定 每个供体的空间分布。RCTD50 使用 spacexr R 包(v.2.2.1)在双重模式 下实现,应用于表达矩阵,使用参考图数据集20中的 MIV2 Velasco 类器 官的 scRNAseq 数据作为参考,将细胞类型权重分配给空间坐标。
报告摘要 有关研究设计的更多信息,请参阅 Nature 与本文相关的投资组合报告摘要。
¶ 代码可用性
在数据分析过程中使用的代码可在GitHub上获取 (https://github.com/tfait s/Arlotta_Lab_Chimeroids)。
38. Church, G. M. The Personal Genome Project. Mol. Syst. Biol. 1, 2005.0030 (2005).
39. Sheridan, S. D. et al. Epigenetic characterization of the FMR1 gene and aberrant neurodevelopment in human induced pluripotent stem cell models of fragile X syndrome. PLoS ONE 6, e26203 (2011).
40. Sugathan, A. et al. CHD8 regulates neurodevelopmental pathways associated with autism spectrum disorder in neural progenitors. Proc. Natl Acad. Sci. USA 111, E4468–E4477 (2014).
41. Thomson, J. A. et al. Embryonic stem cell lines derived from human blastocysts. Science 282, 1145–1147 (1998).
42. Boulting, G. L. et al. A functionally characterized test set of human induced pluripotent stem cells. Nat. Biotechnol. 29, 279–286 (2011).
43. Schindelin, J. et al. Fiji: an open-source platform for biological-image analysis. Nat. Methods 9, 676–682 (2012).
44. Goodchild, S. J. et al. Molecular pharmacology of selective NaV1.6 and dual NaV1.6/ NaV1.2 channel inhibitors that suppress excitatory neuronal activity ex vivo. ACS Chem. Neurosci. 15, 1169–1184 (2024).
45. Zheng, G. X. Y. et al. Massively parallel digital transcriptional profiling of single cells. Nat. Commun. 8, 14049 (2017).
46. Danecek, P. et al. Twelve years of SAMtools and BCFtools. GigaScience 10, giab008 (2021).
47. Chen, Y., Lun, A. T. L. & Smyth, G. K. From reads to genes to pathways: differential expression analysis of RNA-seq experiments using Rsubread and the edgeR quasilikelihood pipeline. F1000Research 5, 1438 (2016).
48. McCarthy, D. J., Chen, Y. & Smyth, G. K. Differential expression analysis of multifactor RNA-Seq experiments with respect to biological variation. Nucleic Acids Res. 40, 4288–4297 (2012).
49. Robinson, M. D., McCarthy, D. J. & Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics 26, 139–140 (2010).
50. Cable, D. M. et al. Robust decomposition of cell type mixtures in spatial transcriptomics. Nat. Biotechnol. 40, 517–526 (2022).




GSEAof uniqueDEGsper donor